ImPACcell
Criblage de molécules bioactives sur lignées cellulaires et larves de poissons zèbres, analyse de leur potentiel thérapeutique ou toxiques par imagerie de fluorescence.

ImPACcell
Criblage de molécules bioactives sur lignées cellulaires et larves de poissons zèbres, analyse de leur potentiel thérapeutique ou toxiques par imagerie de fluorescence.
Imagerie pour analyse du contenu cellulaire (ImPACcell)
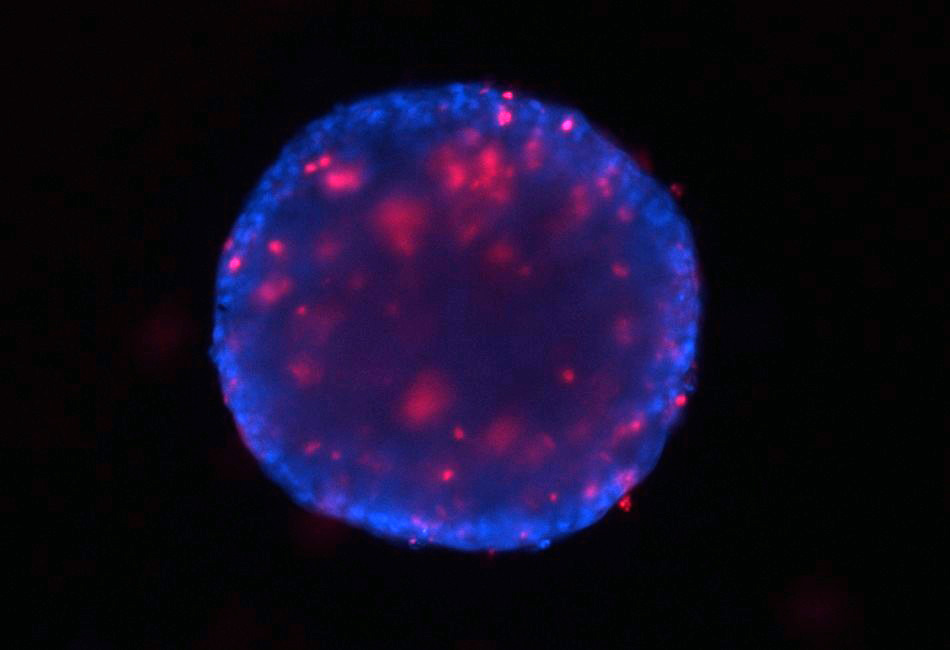
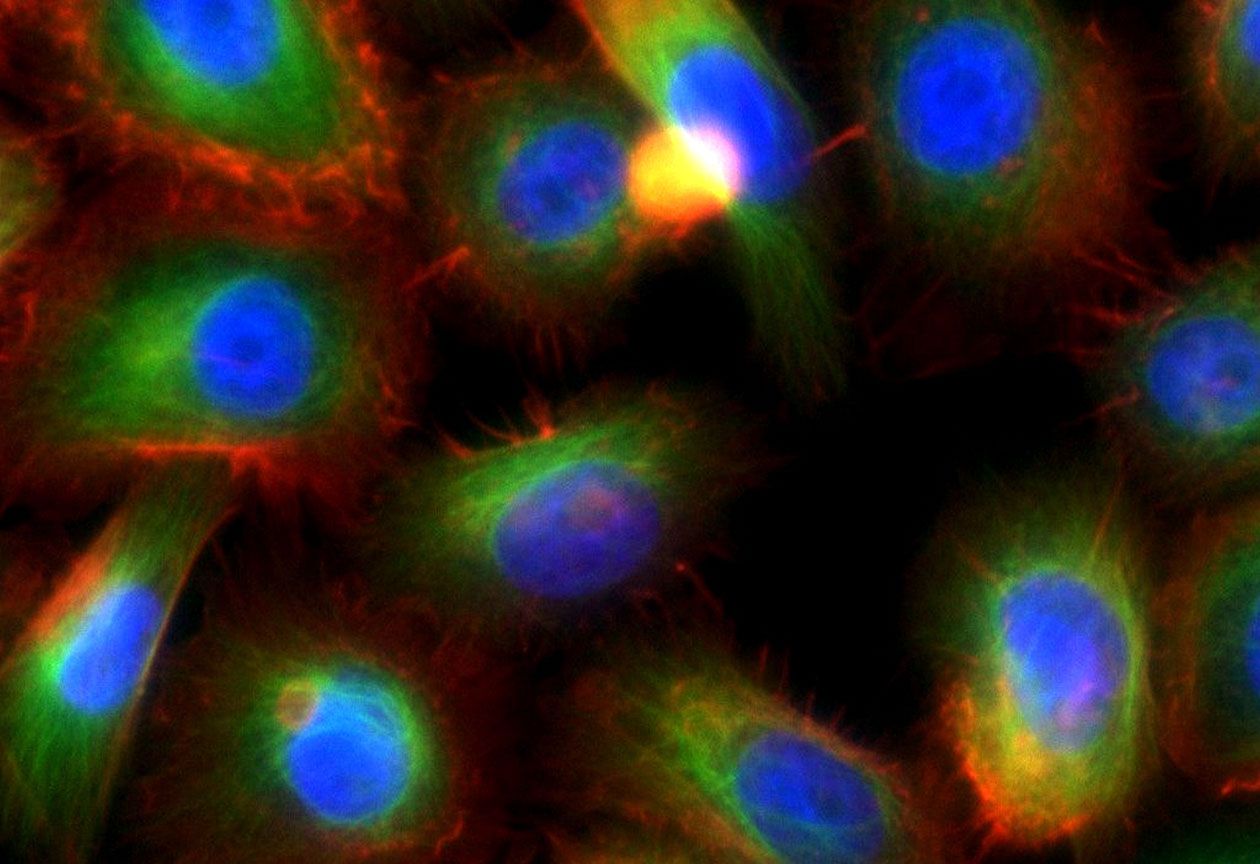
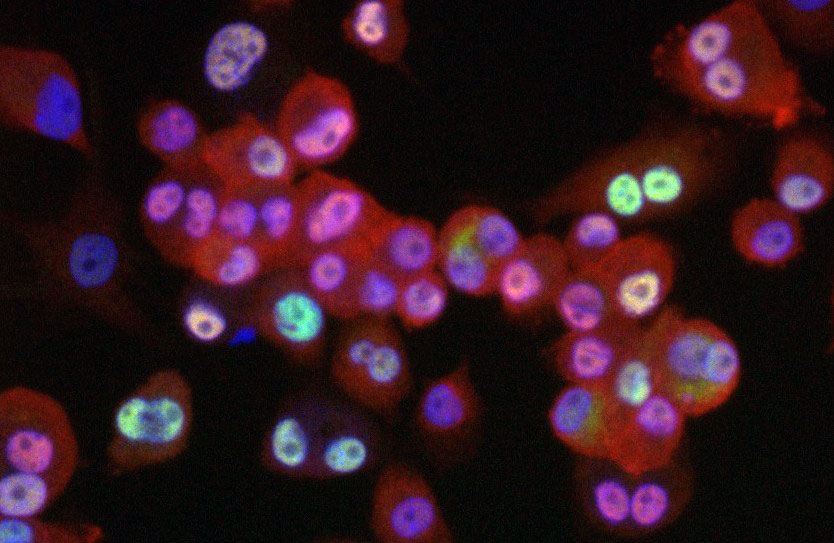
La plateforme ImPACcell réalise des études biologiques à partir d’images de cellules en culture 2D (monocouches) et 3D (sphéroïdes) par microscopie à fluorescence robotisée en HCS (high content screening). Elle propose aussi des tests de criblage HTS (high throughput screening) par bioluminescence ou fluorescence. Un ensemble de tests phénotypiques associés à l’utilisation de 30 lignées cellulaires est disponible afin d’étudier les effets de molécules d’intérêt (chimiques, naturelles, siRNA) ou de facteurs environnementaux (polluants, perturbateurs endocriniens, ondes, vieillissement...) dans des domaines tels que l’environnement, la pharmacologie, la toxicologie, la cosmétologie et le contrôle qualité.
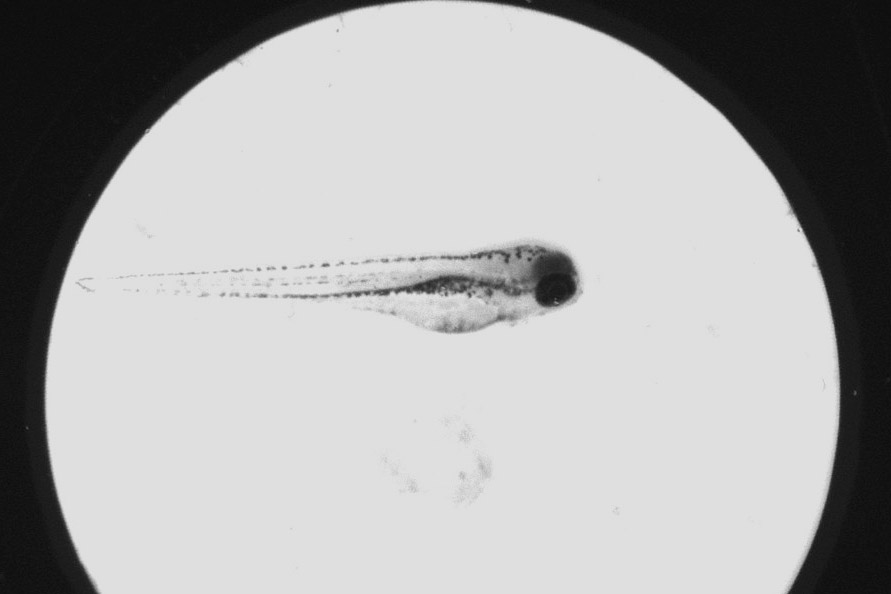
Pour répondre à une forte demande, ImPACcell a récemment mis en place une animalerie poisson zèbre (zebrafish). La plateforme dispose de plusieurs souches transgéniques et sauvages et propose des tests de criblage sur larves (zebratox, angiogenèse, perturbateurs endocriniens...). Cette nouveauté lui permet de développer des tests sur organismes entiers constituant une alternative à l’expérimentation animale d’un point de vue règlementaire.
Expertises et services
- Conduite de projets de criblage biologique de substances toxiques ou thérapeutiques : culture cellulaire, obtention des pontes, marquage en fluorescence, acquisition et analyse d'images, production d’un rapport final,
- Développement et réalisation de tests pour l’analyse de paramètres cellulaires : prolifération, toxicité, cycle cellulaire, stress, immunostimulation, signalisation intracellulaire, dynamique morphologique du cytosquelette, plasticité et motilité cellulaire, angiogenèse, zebratox, polymérisation de la tubuline, production de testostérone...
- Formation et accompagnement à l’utilisation de technologies de criblage,
- Conseil sur les stratégies d’étude des mécanismes biologiques modifiés par l'exposition à des substances variées,
- Participation à des demandes de financements dans le cadre de collaborations.
Moyens et équipements
- Robot d’imagerie HCS ArrayScan XTI (Thermo Scientific, Cellomics) avec acquisition automatisée et analyse en temps réel des images cellulaires,
- Salle de culture L2 entièrement équipée : 3 étuves pour culture cellulaire, poste de sécurité microbiologique (PSM) de niveau II, banque de cellules, robot Hamilton Starlet 8 canaux avec lecteur de codes-barres dédié à la culture cellulaire,
- Robot de distribution, fixation et lavage Hamilton Nimbus 96 canaux avec caisson filtrant,
- Microscope Zeiss inversé pour time lapse,
- Système microfluidique CellASIC ONIX2 et vidéomicroscope Zeiss inversé,
- Système xCELLigence pour l’analyse cellulaire en temps réel sans marquage par mesure d’impédance,
- Fluorimètre et luminomètre SAFAS Xenius pour tests biochimiques et criblage HTS,
- Animalerie zebrafish entièrement équipée : 2 armoires régulées pour l’élevage de poissons zèbres (racks Tecniplast ZebTEC avec active blue technology), 2 incubateurs pour larves de zebrafish, distributeur de nourriture, microscope Zeiss droit à fluorescence et macroscope à fluorescence.









Comment soumettre un projet ?
ImPACcell réalise des tests de criblage, depuis l’incubation des cellules jusqu’à la coloration et l’analyse des résultats. Les utilisateurs peuvent aussi apporter leurs propres préparations cellulaires préalablement fixées et colorées pour l’imagerie et l’analyse HCS.
Pour soumettre un projet, prenez contact avec le responsable technique de la plateforme, Rémy Le Guével. L’équipe d’ImPACcell vous recontactera pour étudier avec vous la faisabilité du projet et la nature des services que la plateforme peut vous offrir. Une réponse vous sera adressée sous huit jours. Si elle est positive, une fiche sera renseignée et vous sera envoyée, avec un devis précisant les tarifs selon votre appartenance (académique local, national, ou entreprise privée).
Les prestations simples nécessitant un protocole en routine seront réalisées dans un délai d’une à deux semaines selon l’occupation de la plateforme. Les projets plus complexes nécessitant l’achat de réactifs ou la mise en culture de cellules particulières seront traités sous trois à huit semaines. Ils peuvent être pris en charge sous la forme de collaborations.
Exemple d'utilisation
Développement d'un test prédictif de la cholestase médicamenteuse
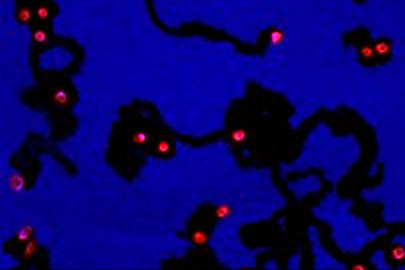
La cholestase intrahépatique est une manifestation fréquente de lésions hépatiques d'origine médicamenteuse. Les mécanismes sous-jacents sont mal connus. ImPACcell a participé au développement d’un test permettant de mettre en évidence des effets cholestatiques sur des cellules HepaRG différenciées en hépatocytes. Adapté aux cellules vivantes en time lapse, ce test offre la possibilité d’étudier en temps réel la dynamique de la modification morphologique et fonctionnelle des canalicules biliaires lors de pathologies hépatiques induites (cholestases).
En parallèle, les experts de la plateforme ont développé un test cellulaire sur cellules HepaRG et une méthode d’analyse HCS permettant la quantification et l’étude morphologique des canalicules biliaires marqués par des sondes spécifiques couplés à des fluochromes. Le développement de ces deux protocoles d’étude, utilisés comme tests prédictifs de la cholestase médicamenteuse, ouvrent de nouvelles perspectives sur les mécanismes conduisant à ces pathologies.
Pour en savoir plus : Sharanek A. et al. (2016). Rho-kinase/myosin light chain kinase pathway plays a key role in the impairment of bile canaliculi dynamics induced by cholestatic drugs. Science Reports, 6(1):24709.
Contact
ImPACcell
Université de Rennes
Bâtiment 8, Campus de Villejean
2 avenue du Pr Léon Bernard
35000 Rennes
Région : Bretagne +33 (0)2 23 23 48 10
+33 (0)2 23 23 48 10
remy.leguevel@univ-rennes1.fr
Site de la plateforme
THÉMATIQUES : Imagerie cellulaire, Imagerie in vivo, radiobiologie
TUTELLES : CNRS, Inserm, Université de Rennes
LABELLISATION IBiSA : 2009
RESPONSABLES SCIENTIFIQUES :
Thierry Charlier
RESPONSABLES TECHNIQUES :
Rémy Le Guével
MOTS CLÉS : Criblage, Toxicité, Thérapeutique, Time lapse, Automatisation, Culture cellulaire, Poisson zèbre, Zebrafish, HCS, HCA, Immunofluorescence
Fiche mise à jour en 2021










