Plateforme de drug discovery Grenoble Alpes (GAP2D)
Criblage, sélection et optimisation de petites molécules pour la biologie et la santé.

Plateforme de drug discovery Grenoble Alpes (GAP2D)
Criblage, sélection et optimisation de petites molécules pour la biologie et la santé.
Plateforme de drug discovery Grenoble Alpes (GAP2D)
La plateforme GAP2D a pour objectif de sélectionner des molécules bioactives afin de les utiliser comme sondes pour la recherche ou comme molécules thérapeutiques. Elle met à disposition des collections de molécules originales et réalise le criblage de chimiothèques à l'aide de tests biologiques sur des protéines purifiées ou des modèles cellulaires. La plateforme fournit également une expertise en chimie médicinale et modélisation moléculaire pour l’optimisation des molécules bioactives sélectionnées.
En recherche, les molécules bioactives sont utilisées pour la détection de phénomènes dynamiques ou l’inhibition d’une fonction biologique de manière dose-dépendante. Elles permettent d'étudier de nouveaux mécanismes cellulaires et de valider des cibles thérapeutiques. En santé, les études de structure-activité (SAR) et de chimie médicinale (MedChem) aident à concevoir de nouvelles molécules thérapeutiques. GAP2D a contribué à la sélection et à l'amélioration de molécules « têtes de série » dans des domaines liés à la santé et la bioénergie.
La plateforme contribue à la formation d'étudiants et de scientifiques à travers l'organisation d'écoles thématiques ou de séminaires. Elle est impliquée dans l'infrastructure nationale ChemBioFrance, les laboratoires d'excellence GRAL et ARCANE, ainsi que dans les programmes EUR et IDEX de l'Université Grenoble Alpes. Elle est partenaire de la Fondation maladies rares (FMR) et du Cancéropôle Lyon Auvergne Rhône-Alpes (CLARA).
Expertises et services
Optimisation et automatisation de tests adaptés au criblage :
- Réalisation de tests en microplaques 96 ou 384 puits et évaluation de la robustesse statistique,
- Réalisation de tests sur cibles purifiées ou sur lysats cellulaires : tests enzymatiques, tests d’interaction protéine-protéine,
- Réalisation de tests phénotypiques adaptés au criblage à haut débit (HTS) sur cellules vivantes ou fixées : viabilité, cytotoxicité, prolifération, apoptose, détection de sondes fluorescentes et biomarqueurs d’intérêt, réponse inflammatoire et stress oxydant,
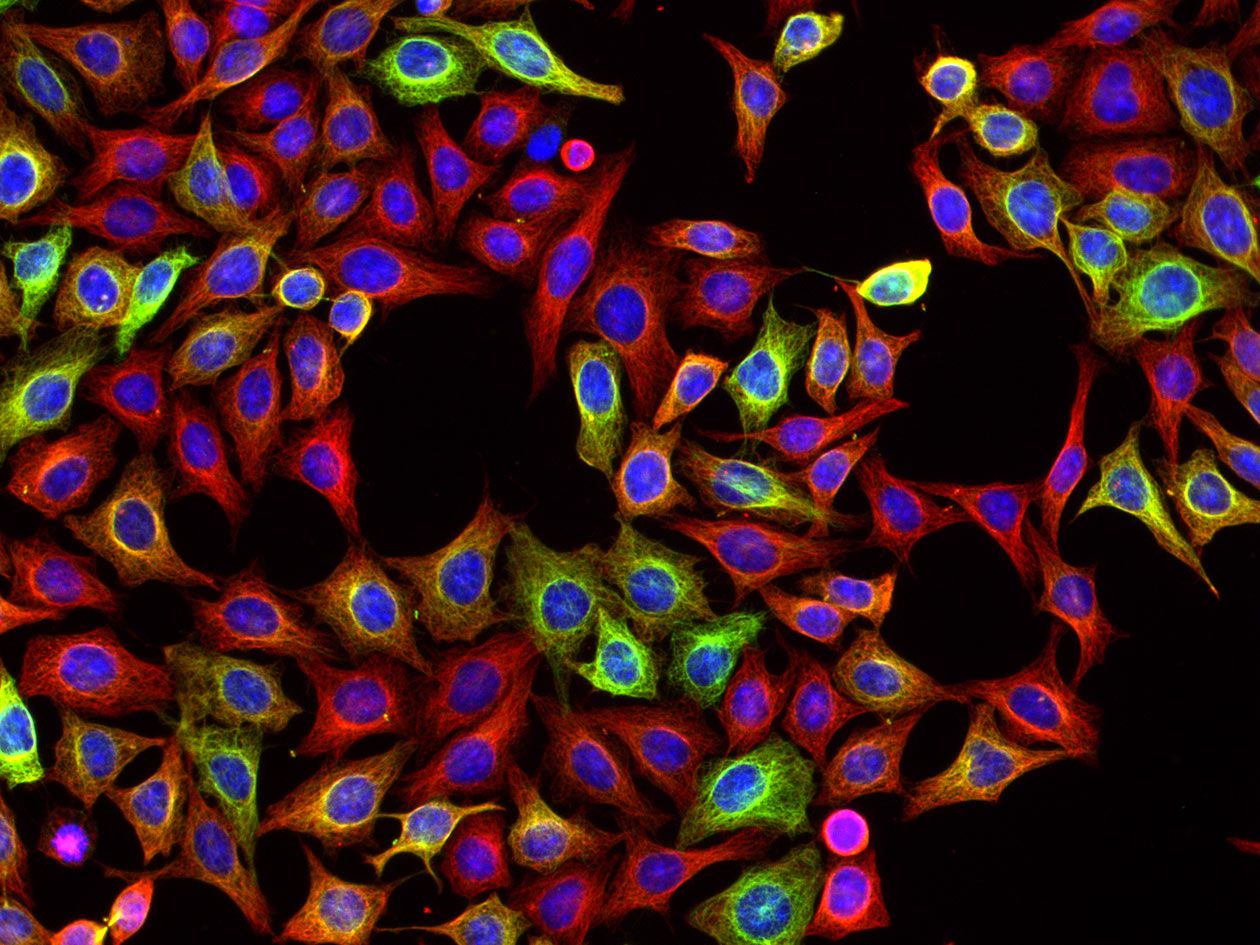
- Réalisation de tests par imagerie automatisée adaptés au criblage à haut contenu (HCS) sur cellules vivantes ou fixées : prolifération, toxicité, apoptose, stress oxydant, autophagie, migration (test de blessure), immunomarquage d’organelles, du cytosquelette cellulaire ou de biomarqueurs d’intérêt, communication intercellulaire, différenciation cellulaire, trafic de récepteurs membranaires, translocation nucléaire de facteurs de transcription.
Criblage à haut débit (HTS) et haut contenu (HCS/HCA) :
- Criblage primaire et sélection de hits,
- Criblage secondaire et validation de hits,
- Réalisation de tests en dose-réponse, de tests de cytotoxicité et de tests orthogonaux.
Profilage de hits ou de collections ciblées :
- Caractérisation de hits sur tests internes adaptés au HTS ou HCS,
- Développement de tests à la demande, y compris sur des modèles cellulaires complexes, sphéroïdes, tumoroïdes et organoïdes.
Chimiothèques :
- Mise à disposition de sous-ensembles de 80 à 70 000 molécules sous contrat de transfert de matériel (MTA) ou libres de propriété intellectuelle,
- Profilage de collections privées.
Chimie médicinale :
- Prédiction de la biodisponibilité et préconisation pour l’optimisation de hits,
- Recherche in silico d'analogues et étude de relations structure-activité (SAR),
- Synthèse chimique (dans des cas spécifiques).
Moyens et équipements
Criblage de molécules :
- Laboratoire de culture cellulaire de niveau 2,
- Criblage à haut débit (HTS) : station de travail EVO 200 (Tecan) entièrement intégrée sous un poste de sécurité microbiologique de niveau 2 et adaptée au criblage sur cellules vivantes, avec têtes de pipetage vers microplaques 96 et 384 puits, incubateur à cellules automatisée, laveur, agitateur orbital, carrousel de stockage, lecteur en absorbance, fluorescence (dont FRET et HTRF) ou luminescence, et lecteurs de codes-barres pour la traçabilité des données,
- Criblage par imagerie automatisée (HCS) : microscopes à fluorescence automatisés CellInsight CX7 LED (Thermo Scientific) et IncuCyte S3 (Sartorius), incubateur à CO2 pour le suivi phénotypique de cellules vivantes sur plusieurs jours et ordinateurs pour la quantification et l’analyse des images.
Chimie médicinale :
- Laboratoires de synthèse et phytochimie organique,
- Espace dédié à la préparation de chimiothèques,
- Equipements : RMN, HPLC-MS, accès aux centres de calcul de l’UMS GRICAD (Grenoble Alpes Recherche, infrastructure de calcul intensif et de données pour la modélisation) et aux équipements lourds (FT-IR, RAMAN, diffraction des rayons X) de la plateforme multiservice de l’Institut de chimie moléculaire de Grenoble (ICMG).





Comment soumettre un projet ?
Si vous êtes intéressé par le criblage de votre propre collection de molécules ou de celles de la plateforme par les technologies HTCS ou HCS, vous pouvez contacter Marie-Odile Fauvarque. Si votre projet concerne l'évaluation des propriétés MedChem de hits ou de molécules d’intérêt thérapeutique, vous pouvez contacter Yung-Sing Wong.
La plateforme GAP2D est ouverte aux partenariats académiques et industriels. Elle intervient sous la forme de collaborations (co-financement, co-authoring et partage de propriété intellectuelle) ou de services (facturation du coût total, propriété intellectuelle au porteur de projet, co-authoring selon accord entre les parties).
Exemple de projet
Sélection d’inhibiteurs de la virulence bactérienne
Pseudomonas aeruginosa est une bactérie pathogène opportuniste qui cause des infections nosocomiales et infecte de manière chronique les patients atteints de mucoviscidose. La plateforme GAP2D a apporté sa collaboration au laboratoire Pathogenèse bactérienne et réponses cellulaires (PBRC) pour la conception d’un test original visant à quantifier les changements morphologiques des cellules lors d'une infection à P. aeruginosa.
Deux collections ciblées d'environ 2 000 composés, comprenant des médicaments approuvés par la FDA et des molécules inspirées de produits naturels synthétisées par GAP2D, ont été criblées en parallèle afin de sélectionner in vitro des molécules perturbant l'assemblage du système de sécrétion de type III, un acteur majeur de la virulence de P. aeruginosa. Des analogues et des molécules chimériques ont été synthétisés à partir de hits, permettant la sélection de molécules « têtes de série » capables de perturber efficacement l’assemblage du système de sécrétion de type III et d’inhiber les dommages cellulaires causés par l’infection à P. aeruginosa, sans affecter la croissance bactérienne in vitro.
Ces pistes sont prometteuses pour le développement d’une stratégie thérapeutique innovante consistant à traiter l’infection en inhibant la virulence de la bactérie, tout en évitant de compromettre la viabilité bactérienne afin de prévenir l’apparition de mécanismes de résistance tels que ceux observés lors des traitements des infections par des antibiotiques.
Pour en savoir plus : Ngo T.-D. et al. (2019). Chimeric protein-protein interface inhibitors allow efficient inhibition of type III secretion machinery and Pseudomonas aeruginosa virulence. ACS Infectious Diseases, 5(11):1843-1854.
Contact
GAP2D
IRIG, CMBA
CEA-Grenoble, bâtiment C3
17 rue des martyrs
38054 Grenoble
Région : Auvergne-Rhône-Alpes +33 (0)4 38 78 50 56
+33 (0)4 38 78 50 56
mofauvarque@cea.fr
Site de la plateforme
THÉMATIQUES : Criblage, chimiothèque, chemobiologie, Imagerie cellulaire
RESPONSABLES SCIENTIFIQUES :
Marie-Odile Fauvarque
RESPONSABLES TECHNIQUES :
Caroline Barette, Emmanuelle Soleilhac, Yung-Sing Wong, Khan-Chi Nguyen-Pham
RESPONSABLES QUALITÉ :
Caroline Barette
TUTELLES : CEA, CNRS, Inserm, Université Grenoble Alpes
INFRASTRUCTURES NATIONALES : ChemBioFrance
LABELLISATION IBiSA : 2018
MOTS CLÉS : Drug discovery, Criblage à haut débit, HTS, Criblage à haut contenu, HCS, Analyse d’images à haut contenu, HCA, Imagerie, Criblage moléculaire, Interaction protéine-protéine, PPI, Profilage de hits, Cribles sur cibles purifiées ou en lysats, Cribles phénotypiques sur cellules vivantes ou fixées, Modèles cellulaires, Sphéroïdes, Optimisation de hits, Chimie médicinale, MedChem,
Fiche mise à jour en 2025










