UCAGenomiX
Analyse quantitative de profils d’expression génique par séquençage courts (Illumina) et longs fragments (Nanopore), de l’organisme entier à la cellule unique.
UCAGenomiX
Analyse quantitative de profils d’expression génique par séquençage courts (Illumina) et longs fragments (Nanopore), de l’organisme entier à la cellule unique.
UCAGenomiX
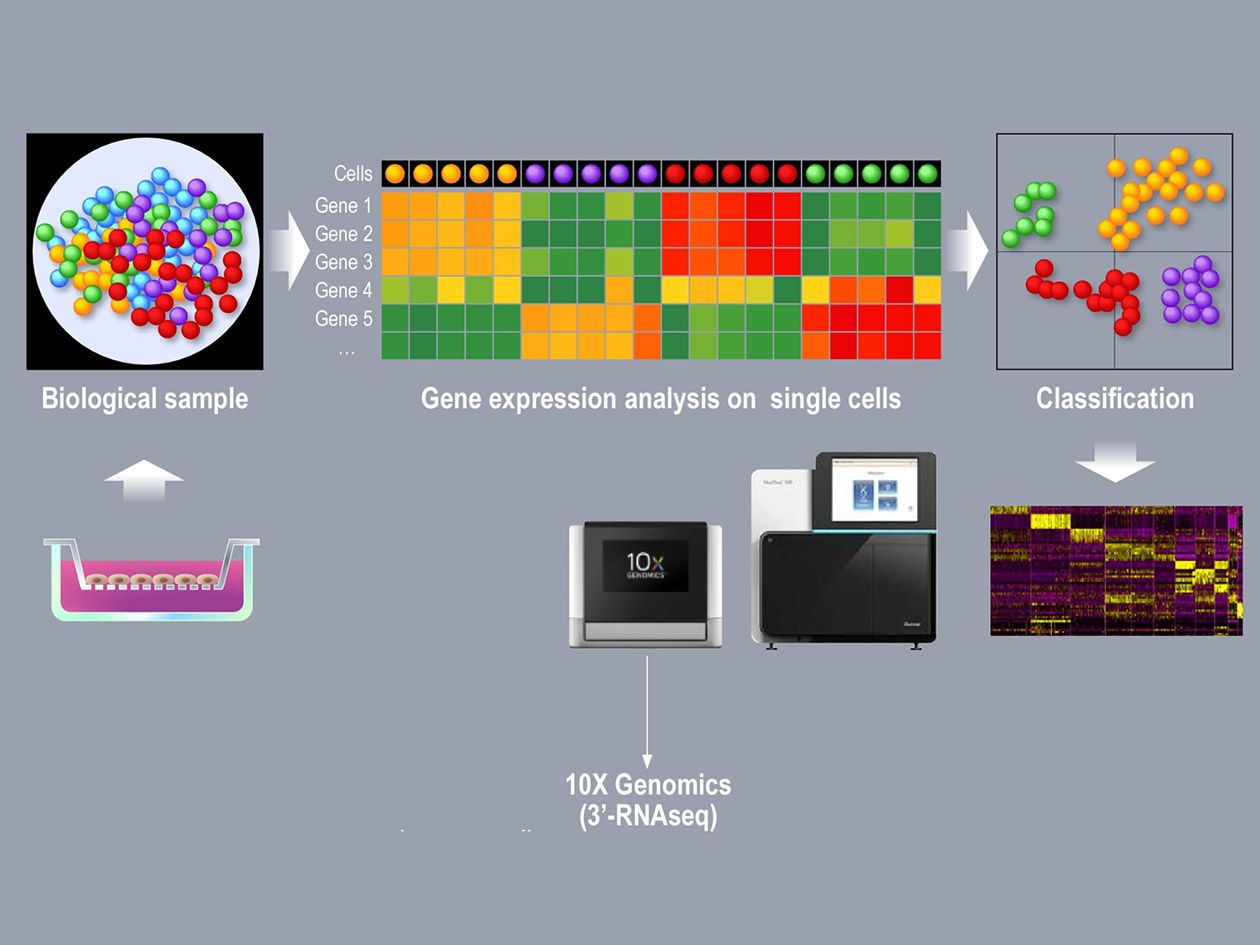
UCAGenomiX propose une expertise en génomique fonctionnelle : conception expérimentale des expériences, analyse bioinformatique primaire et secondaire des résultats, aide à leur interprétation biologique... La plateforme développe en permanence de nouvelles expertises, en interne ou au travers de collaborations scientifiques. Elle met en œuvre tout un panel d’expériences en génomique (DNA-Seq, Chip-Seq, RNA-Seq, CLIP-Seq) avec une spécialisation dans le profilage -omique sur cellule unique (single cell RNA-Seq, CNV-Seq, ATAC-Seq).
Depuis sa création en 1999, la plateforme est associée à de grands projets, comme la création de la ressource franco-britannique de puces à ADN Resogen, issue d’une collaboration entre le Réseau national des génopoles (RNG) et le Conseil de la recherche médicale (MRC) du Royaume-Uni, le projet Human cell atlas (HCA), mené avec le soutien de la Fondation Chan Zuckerberg, et le projet 1011 génomes de levures. UCAGenomiX est membre fondateur de l'infrastructure nationale France Génomique. Ses ingénieurs éclairent les études biologiques en intégrant une image détaillée des évènements génomiques qui leur sont associés.
Expertises et services
- Séquençage à haut-débit sur séquenceurs Illumina NextSeq, Nanopore MinION et PromethION (RNA-Seq, ChIP-Seq, DNA-Seq),
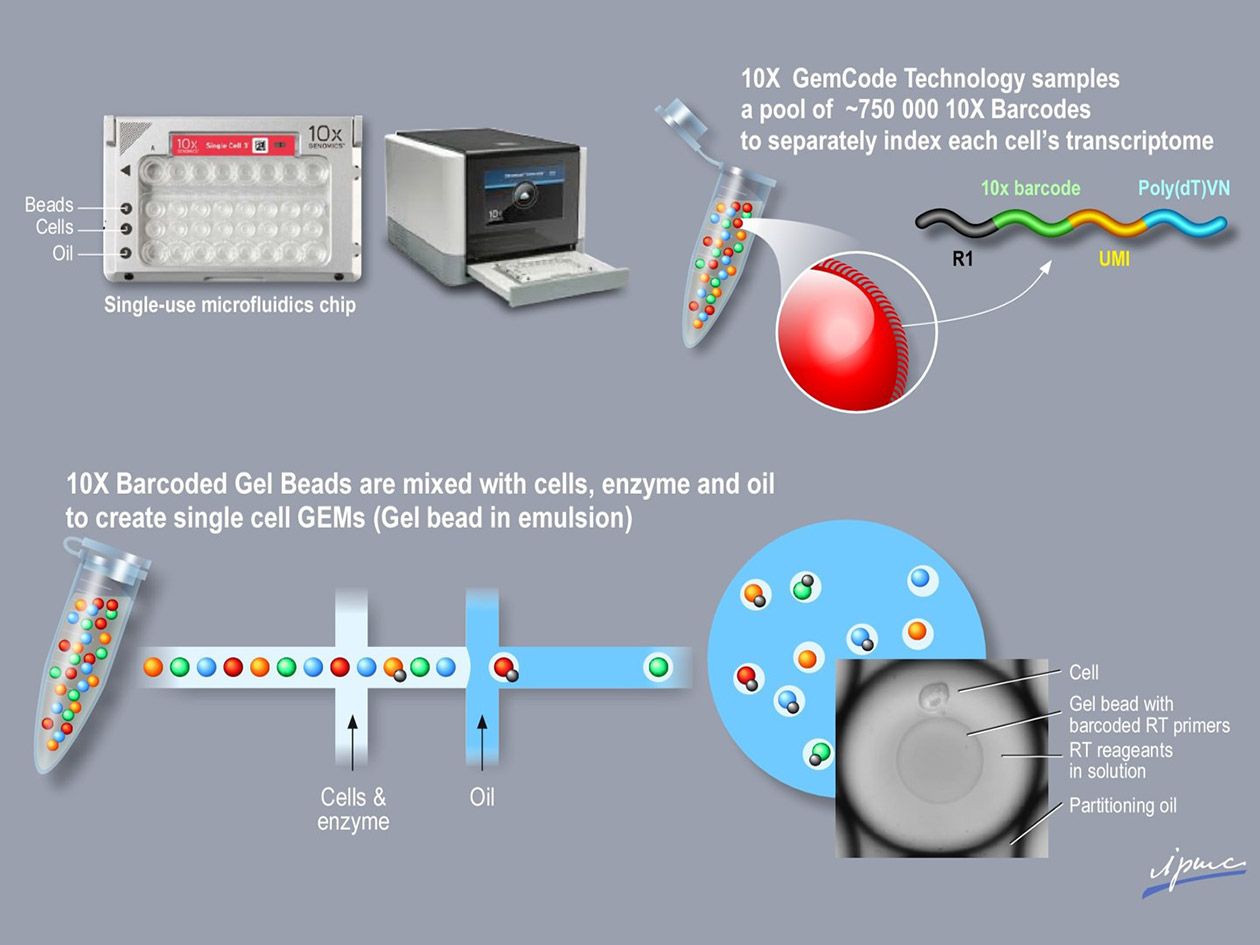
- Profilage d’expression génomique sur cellule unique (single cell) sur la technologie 10x Genomics Chromium (scRNA-Seq, scCNV-Seq, scVDJ-Seq),
- Analyse bioinformatique des expériences conduites sur la plateforme.
Moyens et équipements
- Séquenceur Illumina NextSeq 500,
- Séquenceur Oxford Nanopore Technology PromethION et MinION,
- 10x Genomics Chromium (single cell controller),
- Fluidigm C1 (single cell controller),
- Fluidigm Biomark.
Comment soumettre un projet ?
Les demandes de séquençage à haut-débit doivent être déposées sur le portail de l’infrastructure nationale France Genomique. Elles sont évaluées et validées au cas par cas par le comité scientifique d’UCAGenomiX, puis transférées sur le système d’information de la plateforme, Mediante, qui permet un échange sécurisé des données et des résultats.
Exemple d'utilisation
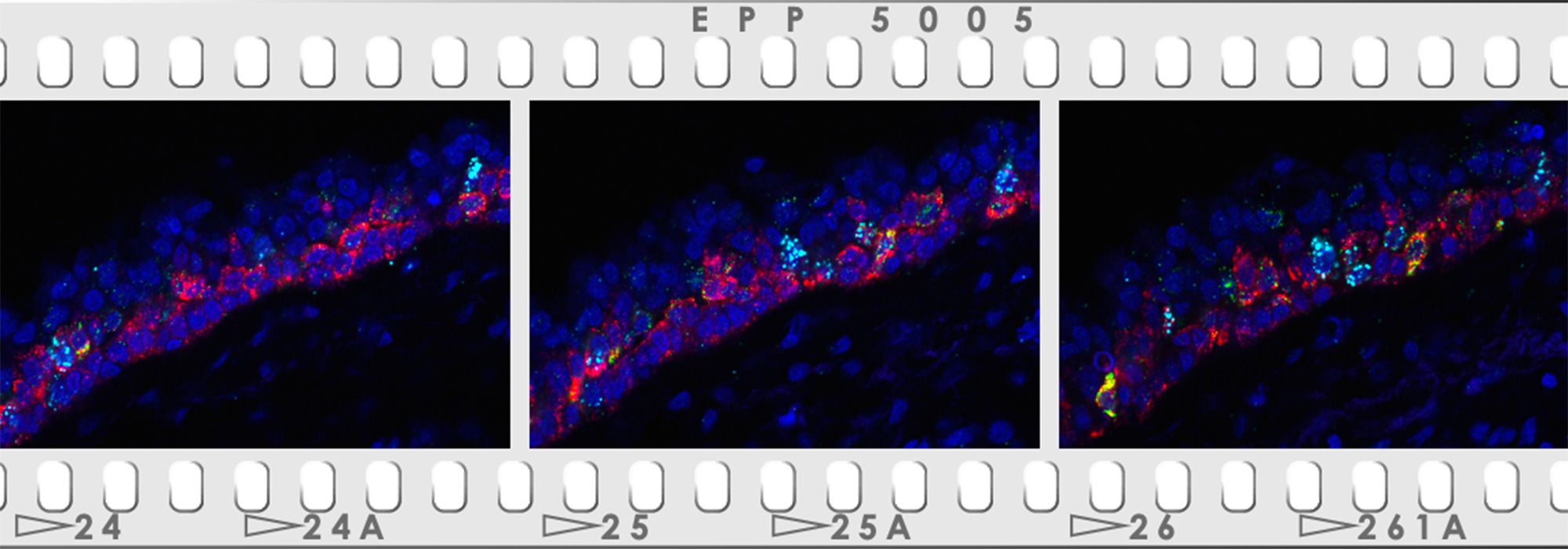
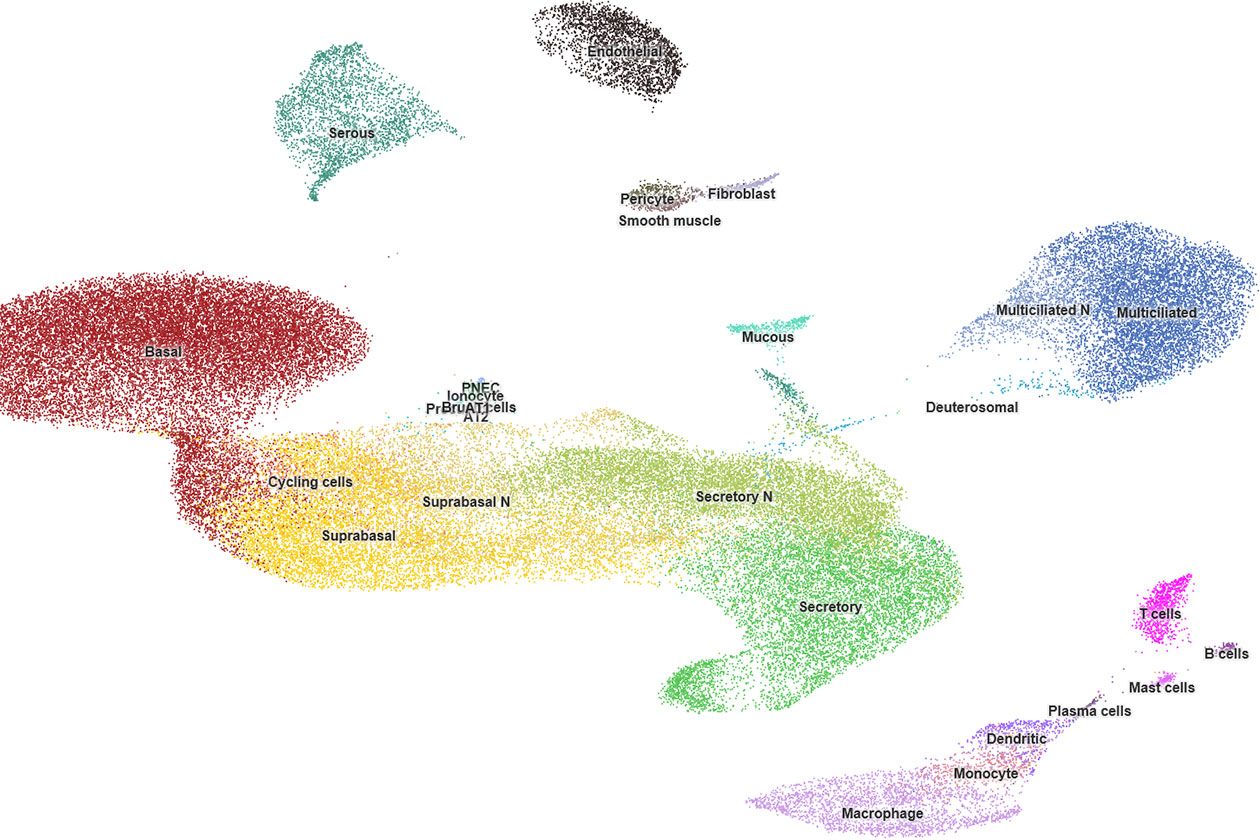
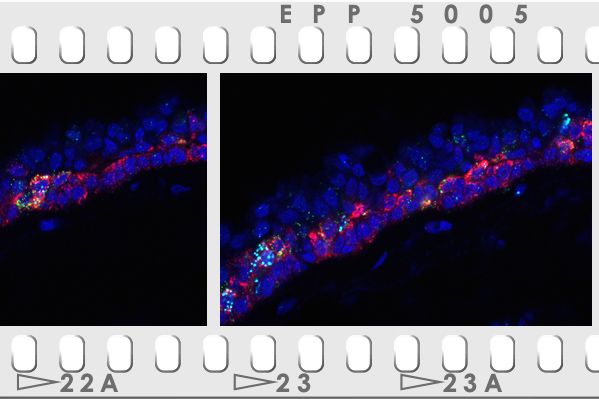
Un atlas d’expression des cellules des voies aériennes supérieures chez l’Homme sain
Les voies respiratoires constituent une ligne de défense établie impliquant des cellules avec des caractéristiques variées. Les méthodes de profilage d’expression sur cellule unique permettent d'étudier la distribution des différentes populations cellulaires mises en jeu et leurs changements en fonction de leur position sur les voies respiratoires.
UCAGenomiX a exploré l'hétérogénéité cellulaire de l'épithélium des voies respiratoires humaines chez 10 volontaires en parfaite santé. 77 969 cellules ont été recueillies par bronchoscopie à 35 endroits distincts, depuis le nez jusqu’à la douzième division de l'arbre respiratoire. L'atlas qui en résulte est composé d'un pourcentage élevé de cellules épithéliales (89,1 %), mais aussi de cellules immunitaires (6,2 %) et stromales (4,7 %) dans des proportions qui varient selon les sites étudiés. Il révèle une expression génétique différentielle entre des types de cellules identiques (cellules suprabasales, sécrétoires ou multiciliées) issues, soit de la zone nasopharyngée (exprimant alors davantage MUC4, PI3, SIX3), soit de la zone trachéobronchique (exprimant alors davantage SCGB1A1 ou TFF3). De façon surprenante, l'expression génique propre à ces mêmes cellules apparait stable parmi l’ensemble des échantillons trachéobronchiques.
Cet atlas améliore la description des ionocytes, des cellules neuroendocrines pulmonaires (PNEC) et des cellules « en brosse », probablement dérivées d'une même population de cellules précurseurs. Une population de cellules KRT13 positives présentant un pourcentage élevé de cellules en division a également été identifié, rappelant les cellules « hillock » déjà décrites chez la souris. La caractérisation robuste de cette grande cohorte de cellules uniques constitue une ressource importante pour de futures études. La description précise du continuum existant de l'épithélium nasal aux divisions successives des voies respiratoires pulmonaires, et le profil d'expression génique stable de ces régions, définissent mieux les conditions dans lesquelles des substituts trachéobronchiques pertinents des maladies respiratoires humaines peuvent être développés. Ces travaux sont en cours de publication.
Contact
UCAGenomiX
Institut de pharmacologie moléculaire et cellulaire (IPMC)
660 route des lucioles
06560 Valbonne Sophia Antipolis
Région : Provence-Alpes-Côte d'Azur +33 (0)4 93 95 77 90
+33 (0)4 93 95 77 90
plateforme@ipmc.cnrs.fr
Site de la plateforme
THÉMATIQUES : Bioinformatique, bases de données, Génomique, transcriptomique
RESPONSABLES SCIENTIFIQUES :
Pascal Barbry
RESPONSABLES TECHNIQUES :
Virginie Magnone, Kevin Lebrigand
RESPONSABLES QUALITÉ :
Géraldine Rios
TUTELLES : CNRS
INFRASTRUCTURES NATIONALES : France Génomique
LABELLISATION IBiSA : 2009
MOTS CLÉS : Séquençage, Cellule unique, Génomique, Transcriptomique, Bioinformatique, RNA-Seq, scRNA-Seq, Single cell, Long reads, Nanopore, PromethION, Illumina, 10x Genomics Chromium
Fiche mise à jour en 2021












