Plateforme protéomique Strasbourg Grand Est (PSGE)
Protéomique fonctionnelle, protéomique structurale, conception et déploiement d’outils bioinformatiques pour la protéomique.

Plateforme protéomique Strasbourg Grand Est (PSGE)
Protéomique fonctionnelle, protéomique structurale, conception et déploiement d’outils bioinformatiques pour la protéomique.
Plateforme protéomique Strasbourg Grand Est (PSGE)
La plateforme PSGE, site ProFi Strasbourg, propose des collaborations et des prestations de service en protéomique fonctionnelle et structurale. Elle s’appuie sur une équipe hautement expérimentée de chercheurs et d’ingénieurs du Laboratoire de spectrométrie de masse bio-organique (LSMBO) de l’Institut pluridisciplinaire Hubert Curien (IPHC).
Le LSMBO mène des activités de R&D en chromatographie, spectrométrie de masse et bioinformatique pour la biologie. Il développe des méthodologies innovantes en protéomique fonctionnelle pour l’identification et la quantification de milliers de protéines, et en protéomique structurale pour la caractérisation de complexes multiprotéiques fonctionnels. Le LSMBO travaille en particulier sur de nouvelles approches quantitatives globales (DIA) et ciblées (PRM), sur l’intégration des multi-omiques (protéogénomique), la spectrométrie structurale et le développement d’outils bioinformatique adaptés à la protéomique.
Expertises et services
- Préparation d’échantillons (gel 1D, 2D, digestion liquide, enrichissement PTMs...),
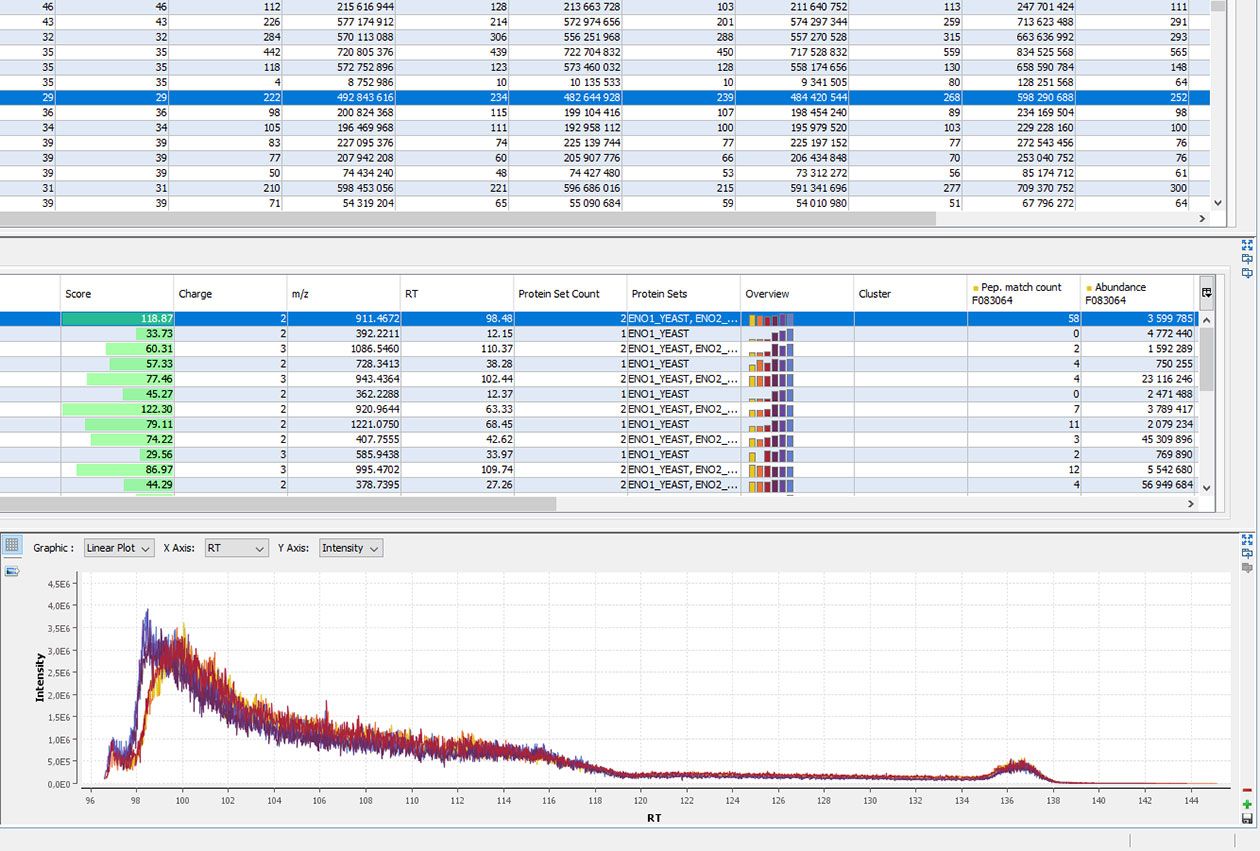
- Identification et quantification globale de protéines à très haut débit (label-free),
- Quantification ciblée de protéines par LC-SRM et LC-PRM,
- Identification et quantification de modifications post-traductionnelles,
- Analyse bioinformatique des données (banques protéiques, séquençage de novo),
- Identification de partenaires de complexes multiprotéiques (interactomique),
- Caractérisation de protéines recombinantes purifiées ou à usage thérapeutique comme les anticorps monoclonaux,
- Analyse structurale de complexes non covalents par MS native et mobilité ionique (complexes multiprotéines, multiprotéines/ADN ou ARN, et protéines/ligand),
- Echange H/D suivis par spectrométrie de masse, epitope mapping,
- Pontage chimique (cross-linking) de complexes multiprotéiques.
Moyens et équipements
- 8 TOF et Q-TOF, 3 Orbitrap, 3 QQQ, HDX Manager, HPLC, UPLC, MALDI-TOF,
- Systèmes nanoLC-MS/MS pour l’analyse protéomique à haut débit : 5 couplages nanoLC-MS/MS, dont 3 Q-TOF et 2 Orbitrap,
- Systèmes microLC-MS/MS pour la quantification par SRM et PRM : 2 couplages micro-LC-QQQ (SRM) et 2 couplages nanoLC-Orbitrap,
- Système nanoLC-MS maXis II (Bruker) avec ETD et MALDI-TOF,
- Instruments pour la MS native et protéomique structurale : 2 systèmes nanoESI-MS native (ESI-TOF pour les hautes masses et Orbitrap EMR pour haute résolution, avec systèmes d’injection automatisée NanoMate Advion), couplage SEC-MS native (Synapt G2, Waters), système HDX-MS (Waters) et 2 couplages de mobilité ionique (Synapt G2Si et G2, Waters),
- Moteurs de recherche, outils bioinformatiques de validation (Proline, Mascot, MaxQuant…) et suites logicielles pour l’interprétation des données de protéomique (Proline et MSDA).







Comment soumettre un projet ?
Pour toute demande de collaboration ou de prestation de service, rendez-vous sur le portail de l’infrastructure nationale de protéomique ProFi. Décrivez brièvement votre projet puis choisissez le site de Strasbourg pour sa réalisation. Le personnel de la plateforme PSGE peut vous accompagner, de la préparation des échantillons à l’interprétation des données. Les prestations réalisées sont gérées dans le cadre d'un système de management de la qualité défini selon la norme ISO 9001 et NF X50-900.
Exemple d'utilisation
Développement d’une méthode de quantification MS Top 3-ID-DIA pour l’analyse in-process des cellules hôtes productrices de médicaments
Au cours des deux dernières décennies, une pléthore d'anticorps thérapeutiques a été approuvée et représente aujourd’hui une voie thérapeutique prometteuse pour de nombreuses pathologies. L'élimination efficace des impuretés protéiques résiduelles de la cellule hôte productrice (HCP), qui peuvent réduire l'efficacité du médicament et/ou induire des réactions indésirables, est une question cruciale dans la production d’anticorps pour la thérapie humaine.
Dans ce contexte, des méthodes alternatives aux dosages immuno-enzymatiques (ELISA) classiquement réalisés sont nécessaires. La plateforme PSGE a développé des méthodes analytiques innovantes de spectrométrie de masse permettant une identification individuelle et une quantification précise de l’ensemble des HCP dans les médicaments finaux ou au cours des étapes du processus de purification. Ces méthodes utilisent en particulier des modes d’acquisition indépendante des données (DIA).
Pour en savoir plus : Husson G. et al. (2018). Dual data-independent acquisition approach combining global HCP profiling and absolute quantification of key impurities during bioprocess development. Analytical Chemistry, 90 (2), 1241-1247.
Contact
PSGE
LSMBO, IPHC
25 rue Becquerel
67087 Strasbourg
Région : Grand Est +33 (0)3 68 85 27 80
+33 (0)3 68 85 27 80
proteomique-sge@unistra.fr
Site de la plateforme
THÉMATIQUES : Bioinformatique, bases de données, Biologie structurale, biophysique, biochimie, Protéomique
RESPONSABLES SCIENTIFIQUES :
Sarah Cianferani
RESPONSABLES TECHNIQUES :
Christine Schaeffer-Reiss
RESPONSABLES QUALITÉ :
Hélène Diemer
TUTELLES : CNRS, Université de Strasbourg
INFRASTRUCTURES NATIONALES : ProFi
LABELLISATION IBiSA : 2009
MOTS CLÉS : Protéomique, Quantification ciblée, Quantification label-free, MS native, HDX-MS, Mobilité ionique, Caractérisation de protéines, Bioinformatique, Statistique
Fiche mise à jour en 2021











