EPITRANS
Développement et exploitation d’outils de génomique (forward et reverse) et d’épigénomique pour l’exploration et la valorisation de la diversité allélique végétale multi-espèces.
EPITRANS
Développement et exploitation d’outils de génomique (forward et reverse) et d’épigénomique pour l’exploration et la valorisation de la diversité allélique végétale multi-espèces.
Epigénomique et recherche translationnelle (EPITRANS)
EPITRANS développe et met à disposition de la communauté des ressources et des outils de génétique à haut débit, forward et reverse, chez les espèces cultivées, notamment lorsque les études traditionnelles de génétiques (transformation) ne peuvent s'appliquer.
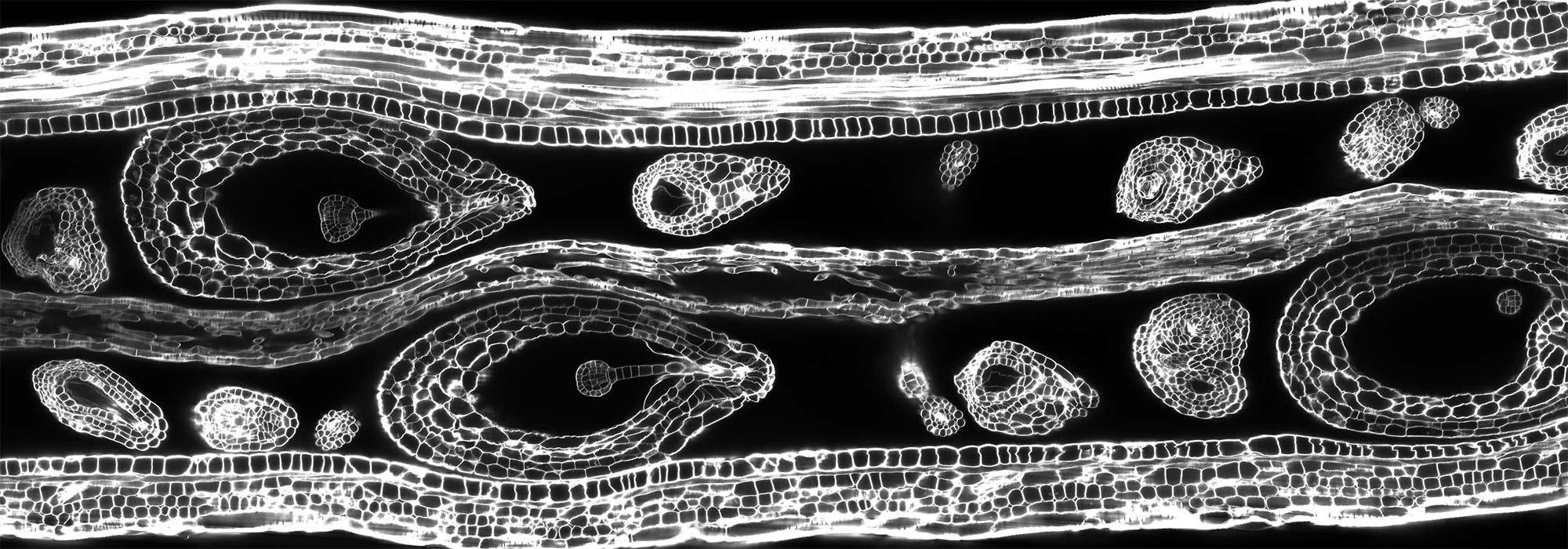
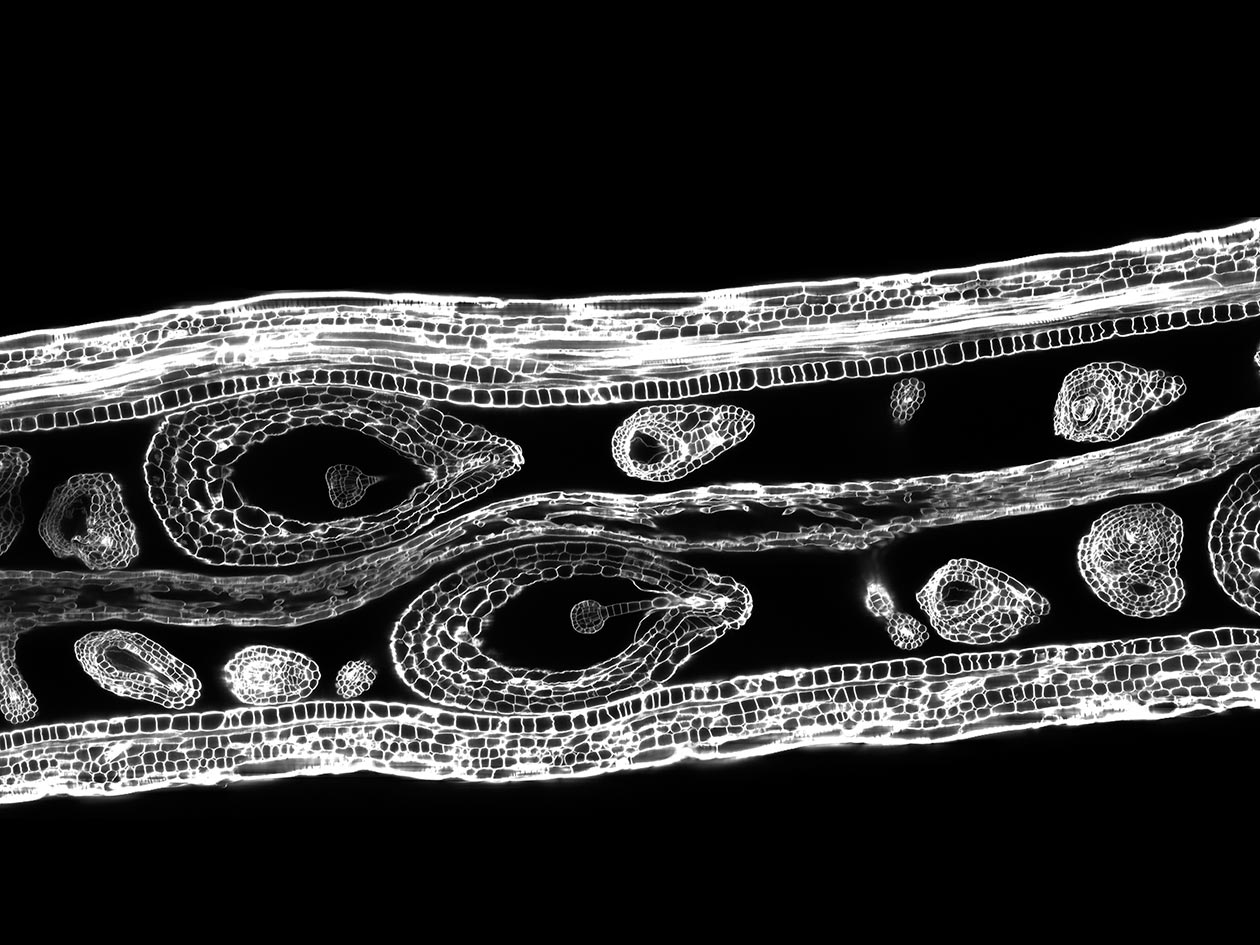
Cinq outils sont ainsi proposés : le clonage positionnel par NGS, le TILLING (targeted induced local lesion in genome) et l'Eco-TILLING, l'épigénomique et le CRISPR. Le premier permet de cloner des gènes d'intérêt agronomique pour ensuite étudier leurs fonctions par TILLING, par recherche de modifications épigénomiques, ou encore par édition du génome via le système CRISPR (selon les espèces). Les allèles découverts, non génétiquement modifiés, sont proposés aux sélectionneurs pour améliorer leurs lignées élites, offrant une alternative aux collections limitées de germoplasmes.
Grâce à la richesse de ses collections de mutants (260 000 lignées) et à la diversité des espèces cultivées disponibles (13 au total), EPITRANS est leader en Europe dans le domaine de la recherche translationnelle. La plateforme a été labellisée infrastructure scientifique collective INRAE.
Expertises et services
Recherche translationnelle :
- Clonage positionnel par séquençage NGS : construction des banques, séquençage et analyse bioinformatique,
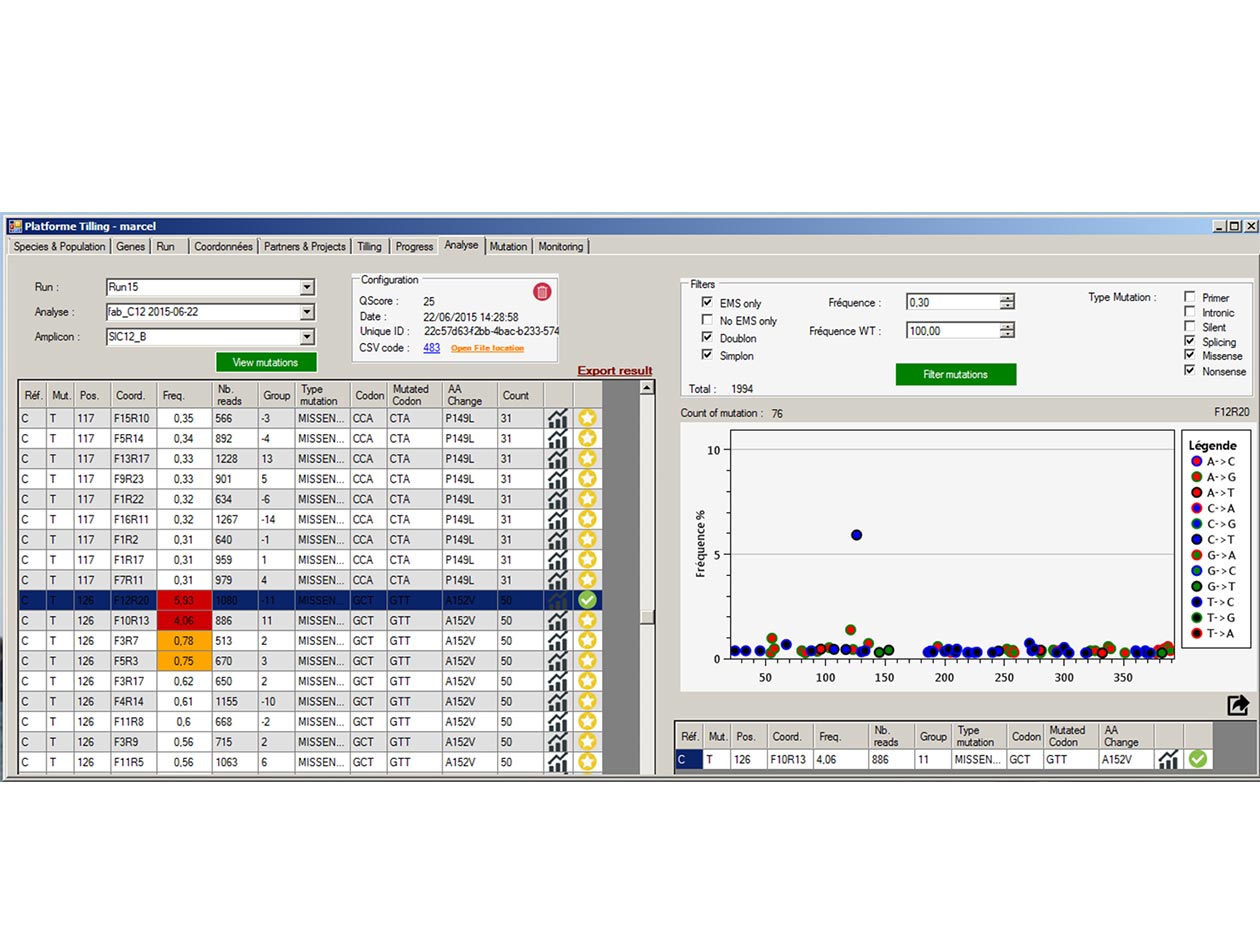
- TILLING et Eco-TILLING par séquençage NGS : production de collections de mutants, construction de banques, séquençage et analyse bioinformatique,
- Mutagenèse saturante : production de collections de mutants saturées et détection de mutations ciblées avec la technologie de PCR digitale en gouttelettes,
- Mutagenèse fast neutron : production de collections de mutants fast neutron et identification de mutants par séquençage NGS et PCR digitale en gouttelettes,
- Edition du génome avec CRISPR-Cas9, notamment dans le cas de familles multigéniques.
Epigénomique à l’échelle du génome :
- Analyse de la méthylation de l'ADN (MeDIP-Seq),
- Analyse des modifications des histones (ChIP-Seq),
- Analyse des sites de liaison des facteurs de transcription (TF-ChIP-Seq),
- Analyse du positionnement des nucléosomes (MNase-Seq),
- Analyse de l'accessibilité de la chromatine (ATAC-Seq),
- Analyse 3D des structures chromatiniques (Hi-C et ChIA-PET)
- Analyse des sites de liaison aux ARNs non-codants (ChIRP-Seq).
Moyens et équipements
- Collections de mutants : 260 000 lignées sur 13 espèces végétales (melon, concombre, pastèque, courgette, calebasse, tomate, poivron, pois, sauge, lin, papaye, Brachypodium et Arabidopsis),
- Extraction d’ADN : lyophilisateur Christ-ISO, station de pipetage automatisée FXp Biomek (Beckman), équipée du module de pression positive pour extraction en filtration,
- Préparation d’échantillons : Chromium Controller (10x Genomics) pour le séquençage de cellules uniques, et microdissecteur laser Arcturus XT LCM (Life Technologies),
- PCR : système de PCR digitale en gouttelettes QX200 et AutoDG (Bio-Rad) et système de PCR en nanovolume Access Array 48.48 IFC (Fluidigm),
- Séquençage : systèmes MiSeq et NextSeq 500 (Illumina),
- Phénotypage : système d’imagerie de plantes in vivo Nightshade LB985 (Berthold Technologies) et transparisateur de tissus X-Clarity (Logos Biosystems).










Comment soumettre un projet ?
Pour soumettre un projet à la plateforme EPITRANS, envoyez un mail à l’adresse contact.epitrans@ips2.upsaclay.fr en précisant dans l’objet du message si celui-ci concerne l'épigénomique ou la biologie translationnelle. Décrivez en quelques lignes votre projet et vos attentes. En fonction de votre demande, la plateforme vous proposera une ou plusieurs options. Au niveau national, elle privilégie les collaborations en partenariat dans le cadre de projets de recherche financés (académiques ou privés). Il est donc recommandé d’anticiper vos demandes. Pour tout autre type de collaboration, EPITRANS est à votre disposition pour établir la meilleure stratégie et satisfaire vos demandes. En cas d’absence de réponse dans le mois qui suit la demande, n’hésitez pas à solliciter de nouveau la plateforme.
Exemple d'utilisation
Décrypter le déterminisme du sexe chez les cucurbitacées pour améliorer la production de semences hybrides
Au début des années 2000, l’équipe d’Abdelhafid Bendahmane à l’Institut des sciences de plantes des Paris-Saclay (IPS2) se passionne pour l’étude des mécanismes moléculaires qui contrôlent le déterminisme du sexe chez les cucurbitacées, trait agronomique d’intérêt pour la production de semences hybrides. Des outils de génomique fonctionnelle à haut-débit comme le clonage positionnel et le TILLING (targeting induced local lesions in genomes) sont développés afin d’identifier et de valider la fonction des gènes impliqués, donnant naissance à la Plateforme de recherche translationnelle, maintenant connue sous le nom d’EPITRANS.
Soutenus par une bourse Advanced Grant de l’ERC en 2013 (projet SEXYPARTH), ces travaux ont abouti au clonage de cinq gènes, brevetés ou en cours de brevetabilité, permettant d’induire chez n’importe quelle variété de cucurbitacées un morphe sexuel au choix : gynoïque, androïque, monoïque ou hermaphrodite. Des plantes mutantes avec des morphes sexuels différents ont ainsi été produites pour diminuer le coût de production de graines hybrides F1 ou améliorer les rendements. Fort de ce succès et de l’intérêt de l’industrie semencière pour ces morphotypes, un projet de maturation porté par la SATT Paris-Saclay a été sélectionné.
Pour en savoir plus : Latrasse D. et al. (2017). The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics Chromatin, 10:22.
Contact
EPITRANS
Bâtiment 630, rue de Noetzlin
Plateau du Moulon
91190 Gif-sur-Yvette
Région : Île-de-France +33 (0)1 69 15 76 75
+33 (0)1 69 15 76 75
marion.dalmais@inrae.fr
Site de la plateforme
THÉMATIQUES : Génomique, transcriptomique
RESPONSABLES SCIENTIFIQUES :
Abdelhafid Bendahmane
RESPONSABLES TECHNIQUES :
Marion Dalmais
TUTELLES : INRAE
LABELLISATION IBiSA : 2010
MOTS CLÉS : Biologie translationnelle, Epigénomique, Plantes cultivées, Clonage positionnel, TILLING, Mutagenèse en saturation, CRISPR-Cas9, Fast neutron, Cellule unique, Mutants, NGS, Dissection laser, Transparisateur de tissus
Fiche mise à jour en 2021










