UCAGenomiX
Analyse quantitative de profils d’expression génique par séquençage courts (Illumina) et longs fragments (Nanopore), de l’organisme entier à la cellule unique.
UCAGenomiX
Analyse quantitative de profils d’expression génique par séquençage courts (Illumina) et longs fragments (Nanopore), de l’organisme entier à la cellule unique.
UCAGenomiX
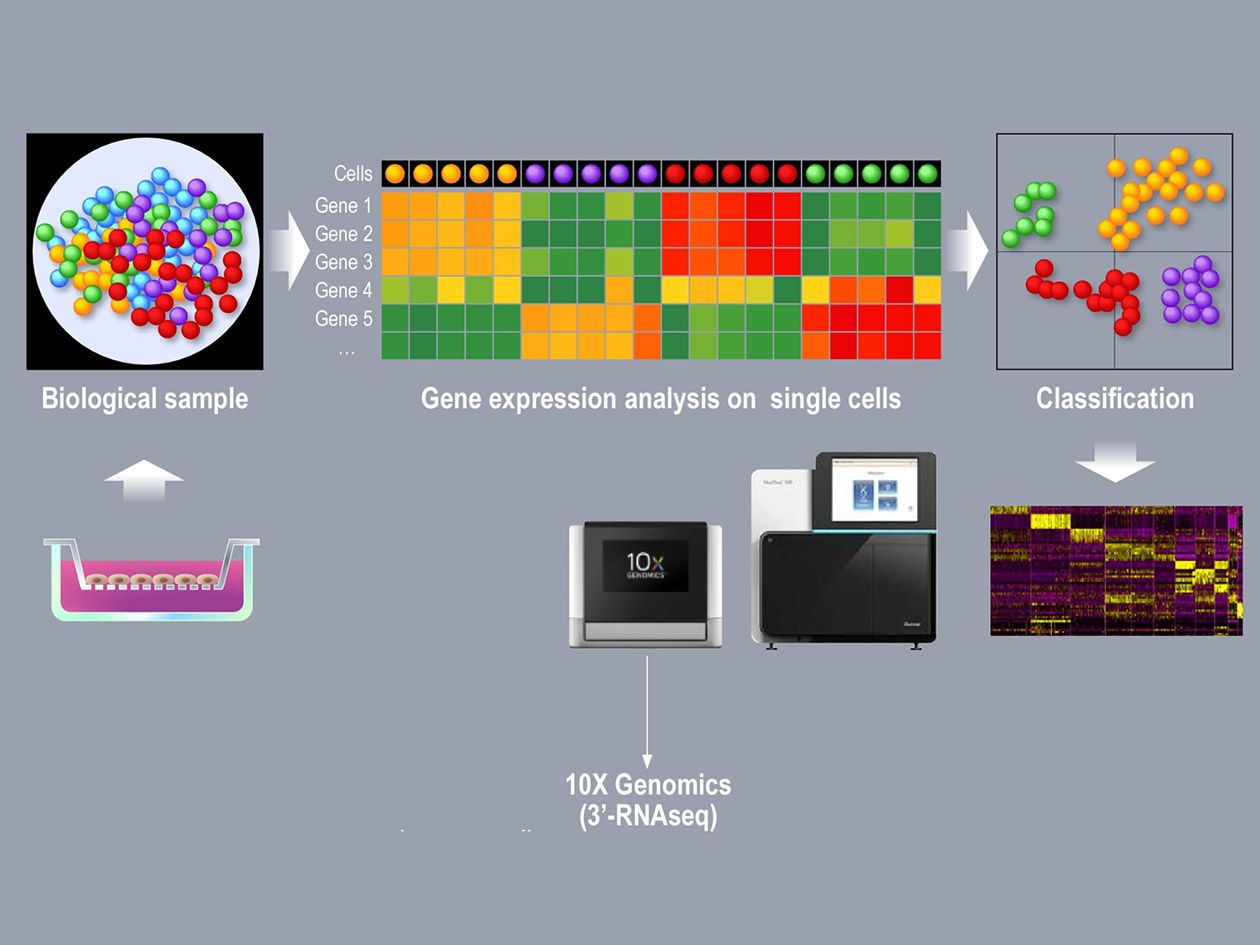
La plateforme UCA GenomiX a été créée en 1999. Initialement orientée vers la conception, la fabrication et l’analyse de puces à ADN, elle a contribué à populariser cette nouvelle technologie en rendant opérationnels dans un environnement certifié ISO9001 et NFX50-900 des outils biotechnologiques de pointe et un système d’information qui ont permis la production et la gestion de grandes masses de données sur plus de 20 ans. L’activité de la plateforme est actuellement centrée sur le séquençage à haut débit des acides nucléiques. UCA GenomiX dispose notamment d’une capacité importante pour traiter de grandes collections d’échantillons, y compris à l’échelle de la cellule unique et par transcriptomique spatiale.
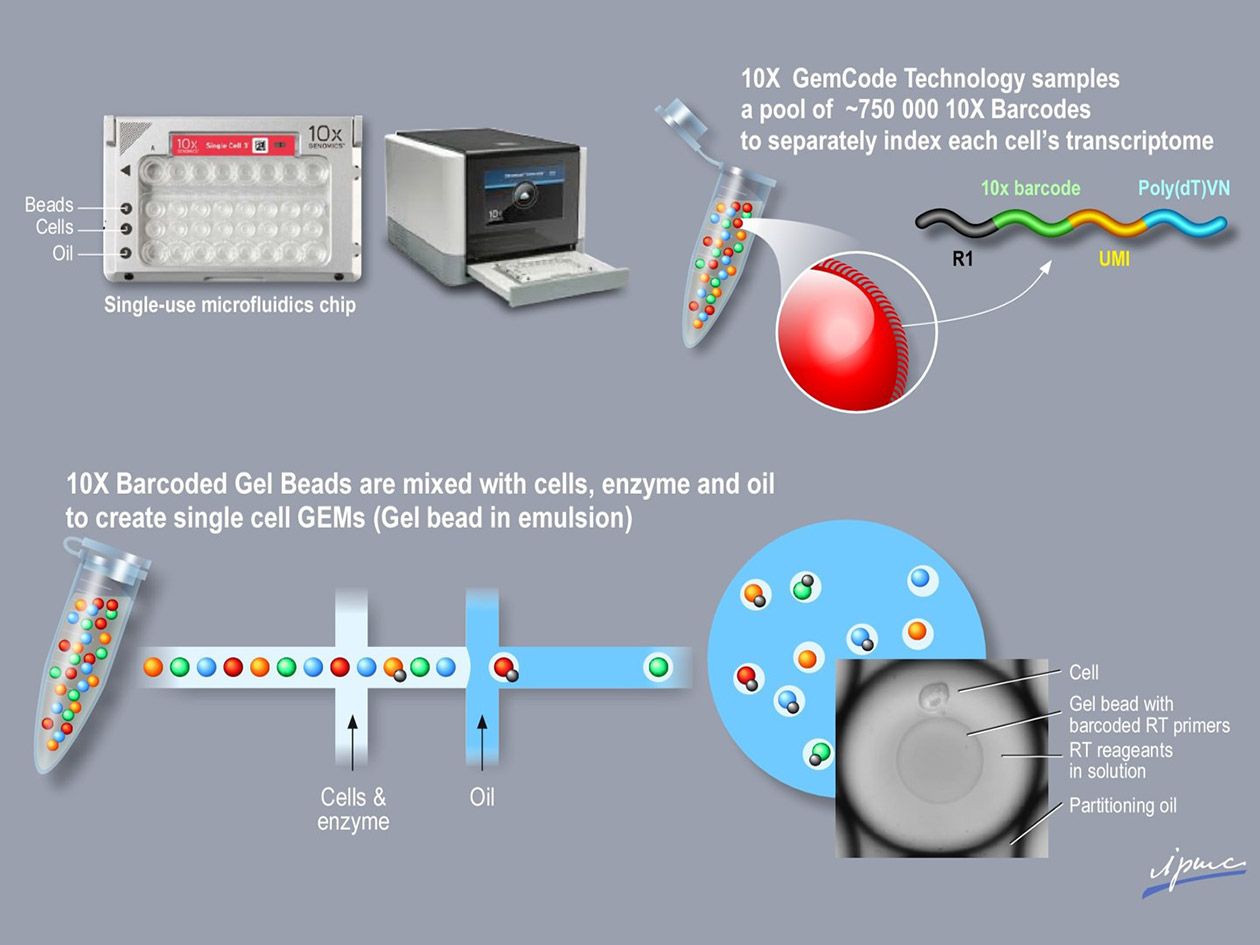
L’activité de routine de la plateforme repose sur un séquenceur à courtes lectures NextSeq 2000 Illumina et un séquenceur à longues lectures PromethION Nanopore, tous deux ouverts à des séquençages d’ADN et d’ARN. Le profilage d’expression génique sur cellule ou noyau unique est réalisable grâce à des technologies Chromium 10x Genomics et Parse Bioscience (scRNA-Seq, scVDJ-Seq, scATAC-Seq…). La plateforme est pionnière dans le séquençage d'ARN en lecture longue à la résolution de la cellule unique (ScNaUmi-Seq, SiCeLoRe), ou spatiale (SiT). Elle dispose enfin des équipements Merscope Vizgen et Xenium 10x Genomics pour réaliser des analyses en transcriptomique spatiale permettant d’étudier jusqu’à 5 000 gènes en parallèle.
Expertises et services
- Séquençage à haut-débit sur séquenceurs NextSeq Illumina, MinION et PromethION Nanopore (RNA-Seq, DNA-Seq),
- Profilage d’expression génomique en single cell sur les technologies Chromium 10x Genomics et Parse Bioscience (scRNA-Seq, scATAC-Seq, scVDJ-Seq),
- Transcriptomique spatiale sur les technologies Xenium et Visium HD 10x Genomics, ou Merscope Vizgen,
- Analyse bioinformatique des expériences conduites sur la plateforme.
Moyens et équipements
- Pré-séquençage : Nanodrop, Bioanalyzer, Qubit et Blue Pippin,
- Séquençage : NextSeq 2000 Illumina, MinION et PromethION Oxford Nanopore,
- Analyse en single cell : Chromium X 10x Genomics, Parse Bioscience et FLASH-Seq (UMI-Single Cell, UMI-Bulk),
- Transcriptomique spatiale : Merscope Vizgen et Xenium 10x Genomics.
Comment soumettre un projet ?
Les demandes de séquençage à haut-débit doivent être déposées sur le portail de l’infrastructure nationale France Genomique. Elles sont évaluées et validées au cas par cas par le comité scientifique d’UCAGenomiX, puis transférées sur le système d’information de la plateforme, Mediante, qui permet un échange sécurisé des données et des résultats.
Exemple de projet
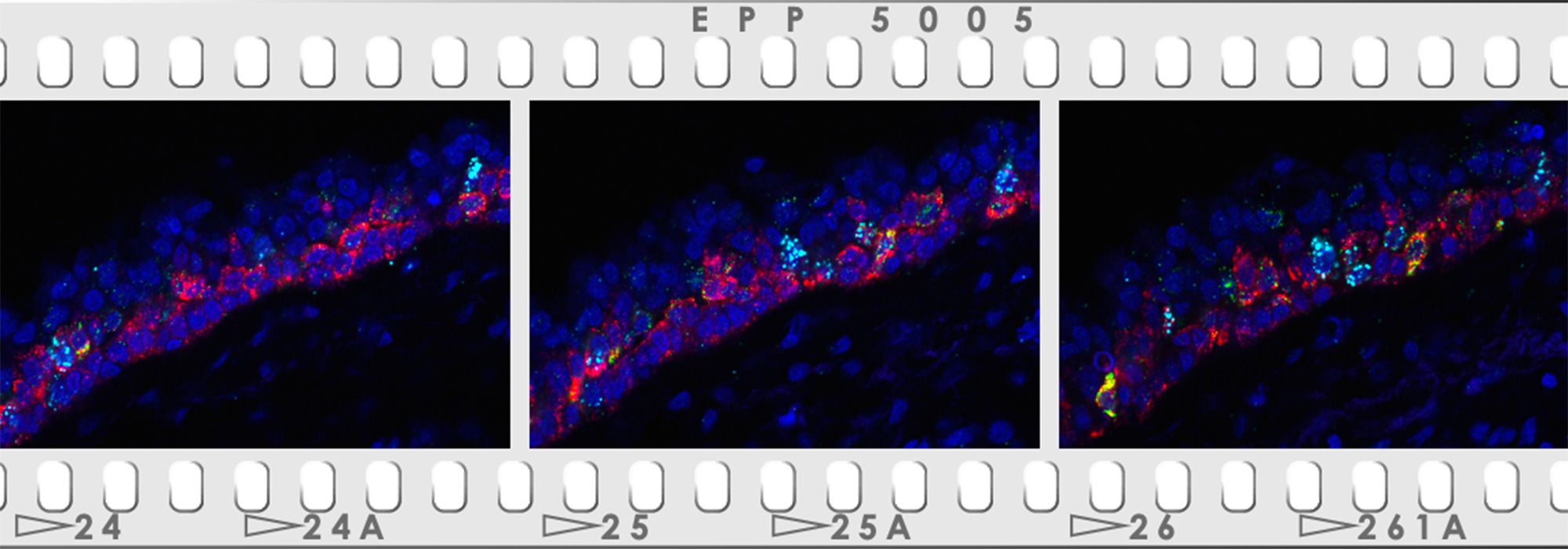
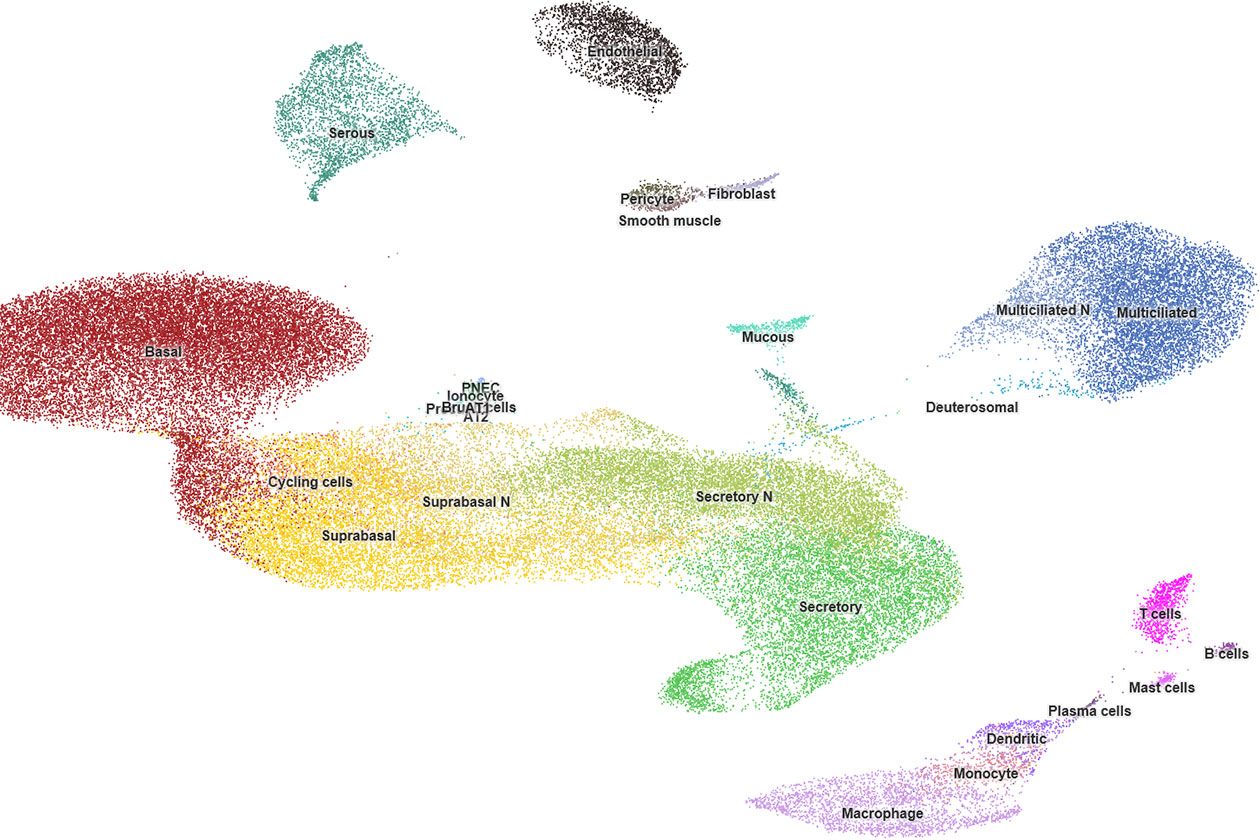
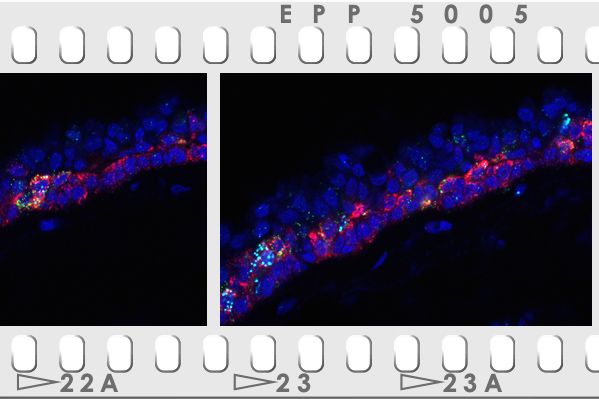
Un atlas d’expression des cellules des voies aériennes supérieures chez l’Homme sain
Les voies respiratoires constituent une ligne de défense établie impliquant des cellules avec des caractéristiques variées. Les méthodes de profilage d’expression sur cellule unique permettent d'étudier la distribution des différentes populations cellulaires mises en jeu et leurs changements en fonction de leur position sur les voies respiratoires.
UCAGenomiX a exploré l'hétérogénéité cellulaire de l'épithélium des voies respiratoires humaines chez 10 volontaires en parfaite santé. 77 969 cellules ont été recueillies par bronchoscopie à 35 endroits distincts, depuis le nez jusqu’à la douzième division de l'arbre respiratoire. L'atlas qui en résulte est composé d'un pourcentage élevé de cellules épithéliales (89,1 %), mais aussi de cellules immunitaires (6,2 %) et stromales (4,7 %) dans des proportions qui varient selon les sites étudiés. Il révèle une expression génétique différentielle entre des types de cellules identiques (cellules suprabasales, sécrétoires ou multiciliées) issues, soit de la zone nasopharyngée (exprimant alors davantage MUC4, PI3, SIX3), soit de la zone trachéobronchique (exprimant alors davantage SCGB1A1 ou TFF3). De façon surprenante, l'expression génique propre à ces mêmes cellules apparait stable parmi l’ensemble des échantillons trachéobronchiques.
Cet atlas améliore la description des ionocytes, des cellules neuroendocrines pulmonaires (PNEC) et des cellules « en brosse », probablement dérivées d'une même population de cellules précurseurs. Une population de cellules KRT13 positives présentant un pourcentage élevé de cellules en division a également été identifié, rappelant les cellules « hillock » déjà décrites chez la souris. La caractérisation robuste de cette grande cohorte de cellules uniques constitue une ressource importante pour de futures études. La description précise du continuum existant de l'épithélium nasal aux divisions successives des voies respiratoires pulmonaires, et le profil d'expression génique stable de ces régions, définissent mieux les conditions dans lesquelles des substituts trachéobronchiques pertinents des maladies respiratoires humaines peuvent être développés. Ces résultats ont fait l'objet d'une publication.
Pour en savoir plus : Deprez M. et al. (2020). A single-cell atlas of the human healthy airways. American Journal of Respiratory and Critical Care Medicine, 202(12):1636-1645.
Contact
UCAGenomiX
Institut de pharmacologie moléculaire et cellulaire (IPMC)
660 route des lucioles
06560 Valbonne Sophia Antipolis
Région : Provence-Alpes-Côte d'Azur +33 (0)4 93 95 77 90
+33 (0)4 93 95 77 90
plateforme@ipmc.cnrs.fr
Site de la plateforme
THÉMATIQUES : Bioinformatique, bases de données, Génomique, transcriptomique
RESPONSABLES SCIENTIFIQUES :
Pascal Barbry
RESPONSABLES TECHNIQUES :
Virginie Magnone, Kevin Lebrigand
RESPONSABLES QUALITÉ :
Géraldine Rios
TUTELLES : CNRS
INFRASTRUCTURES NATIONALES : France Génomique
LABELLISATION IBiSA : 2009
MOTS CLÉS : Séquençage, Cellule unique, Génomique, Transcriptomique, Bioinformatique, RNA-Seq, scRNA-Seq, Single cell, Long reads, Nanopore, PromethION, Illumina, Chronium 10x Genomics
Fiche mise à jour en 2025












