OrganOmics
Un savoir-faire unique en protéomique spatiale, imagerie par spectrométrie de masse et création de modèles cellulaires en 3D pour la biologie et la recherche clinique.
OrganOmics
Un savoir-faire unique en protéomique spatiale, imagerie par spectrométrie de masse et création de modèles cellulaires en 3D pour la biologie et la recherche clinique.
OrganOmics
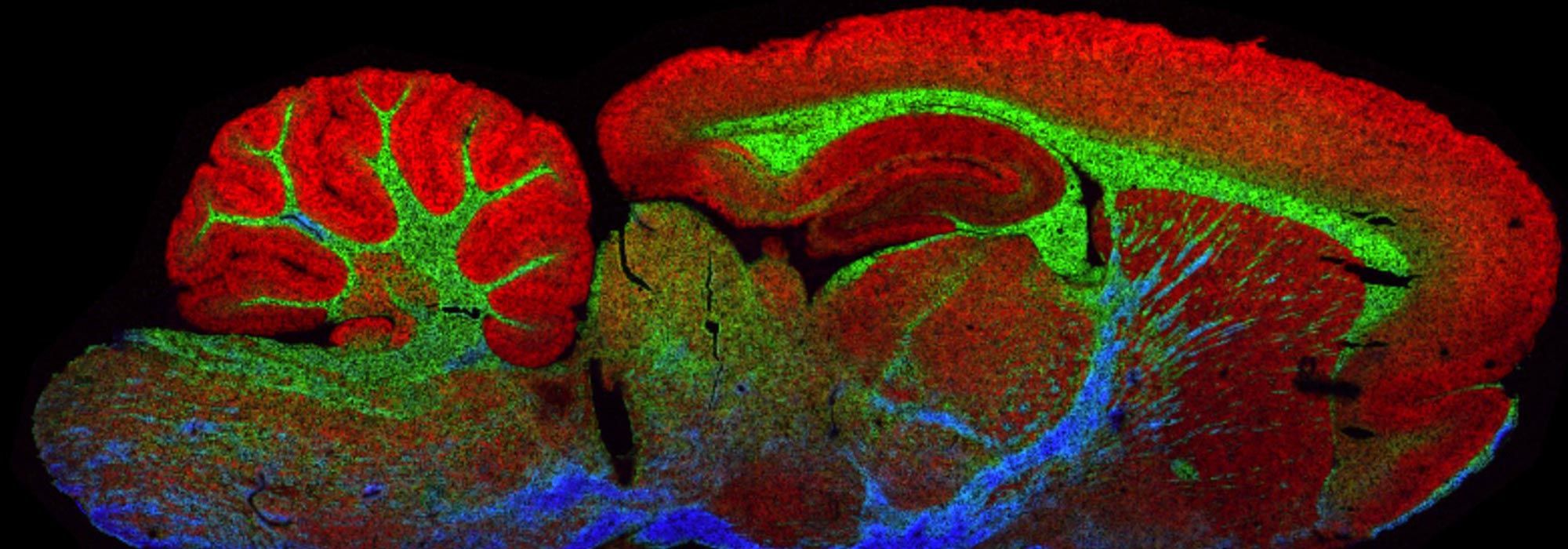
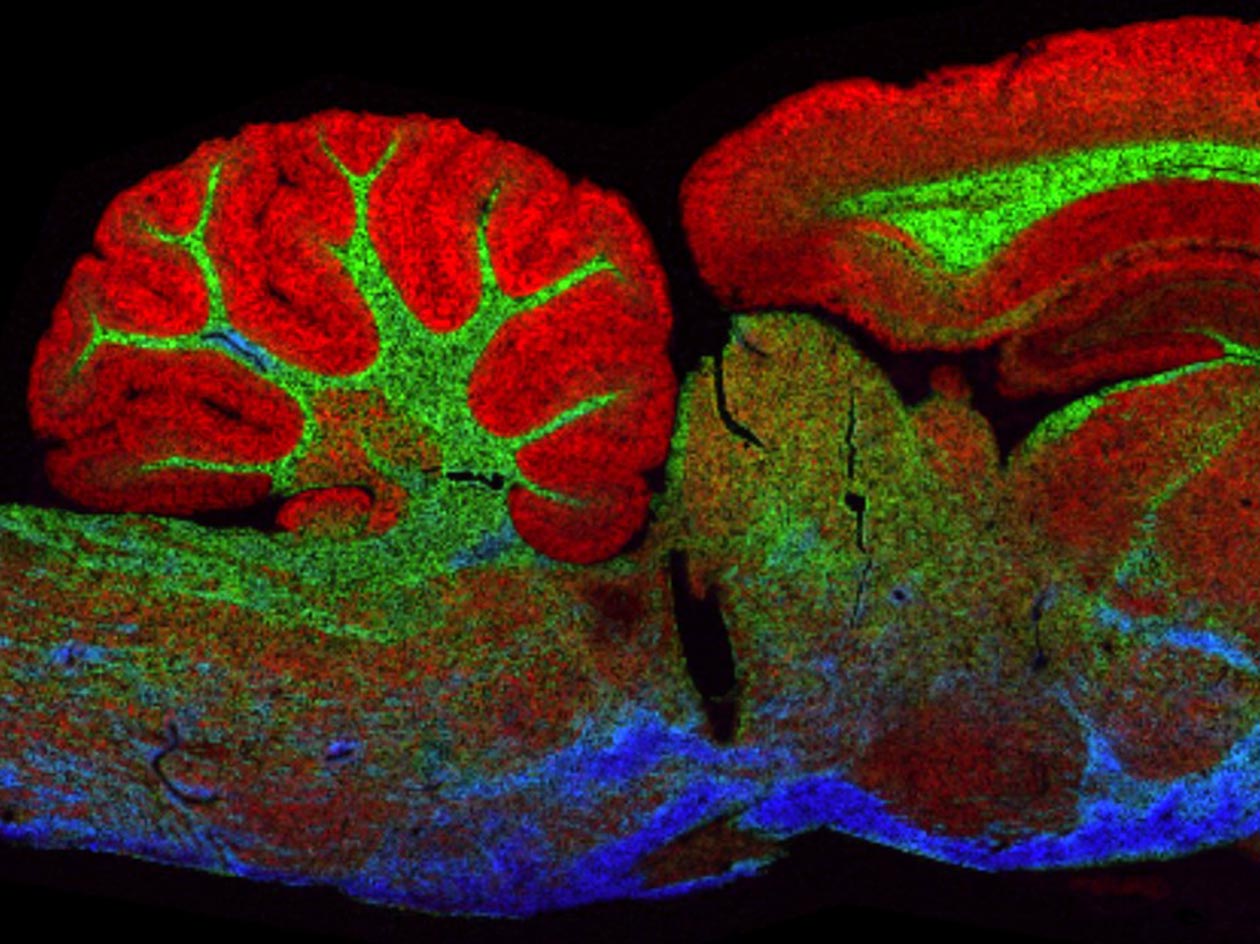
La plateforme OrganOmics détient un savoir-faire unique dans le domaine de la protéomique spatiale, de l’interactomique, de la cellule unique, de l’imagerie par spectrométrie de masse (MS), de la paléoprotéomique et dans la création de modèles cellulaires en 3D (sphéroïdes, organoïdes et bio-impression). Elle est sous-divisée en deux pôles, MS4Omics pour la partie protéomique et 3DCellOmics pour la partie modèles cellulaires.
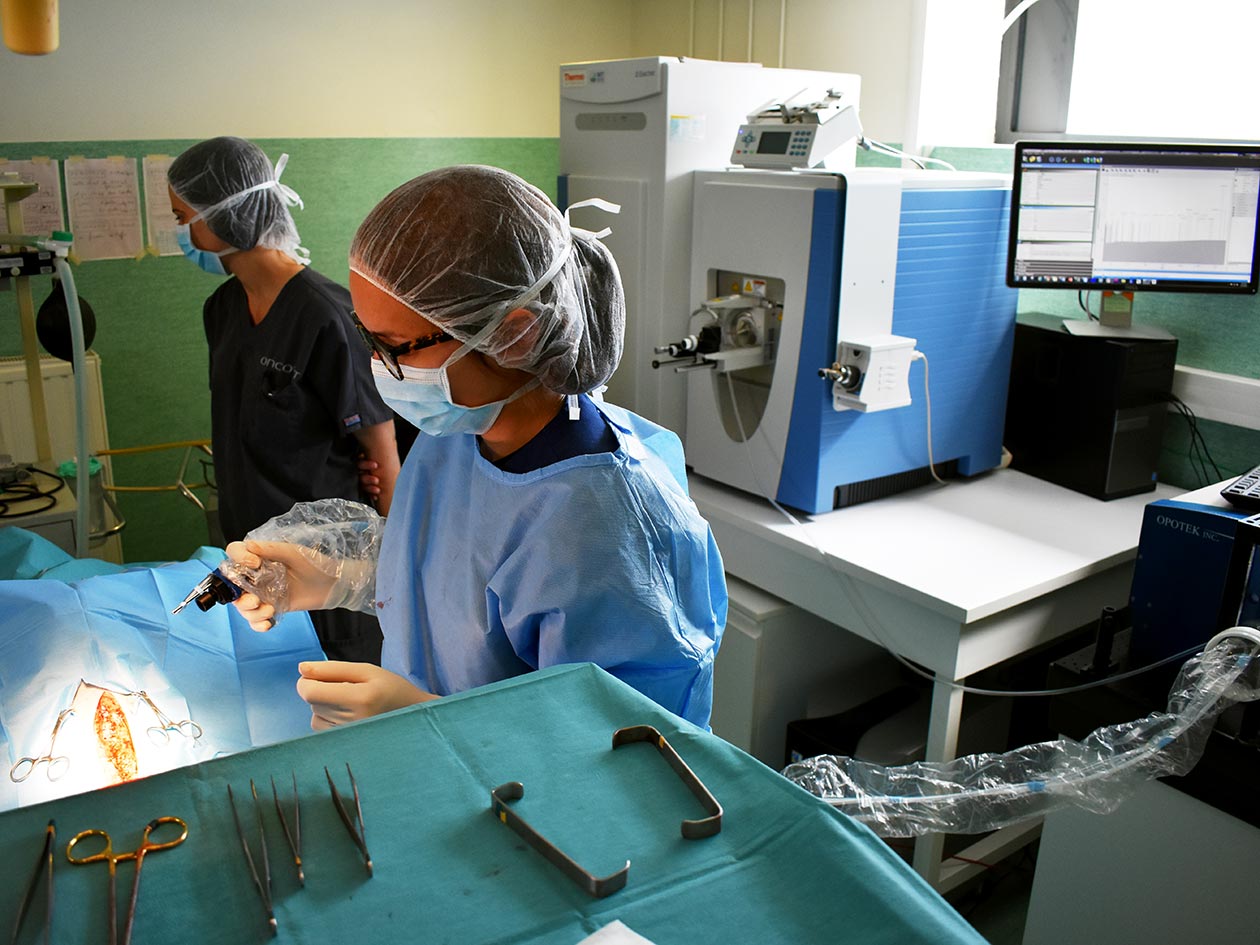
La plateforme est ouverte aux laboratoires académiques, cliniques et industriels, à qui elle propose une large gamme d’analyses et une expertise de haut niveau. Elle dispose en effet d’une grande expérience et d’instruments de dernière génération pour la préparation d’échantillons, l’imagerie MS, la protéomique spatiale et l’analyse de données. Actuellement, elle oriente ses développements vers des approches innovantes pour l’analyse tissulaire à une résolution cellulaire et la conception de modèles 3D de tumeurs contenant des cellules immunitaires.
Expertises et services
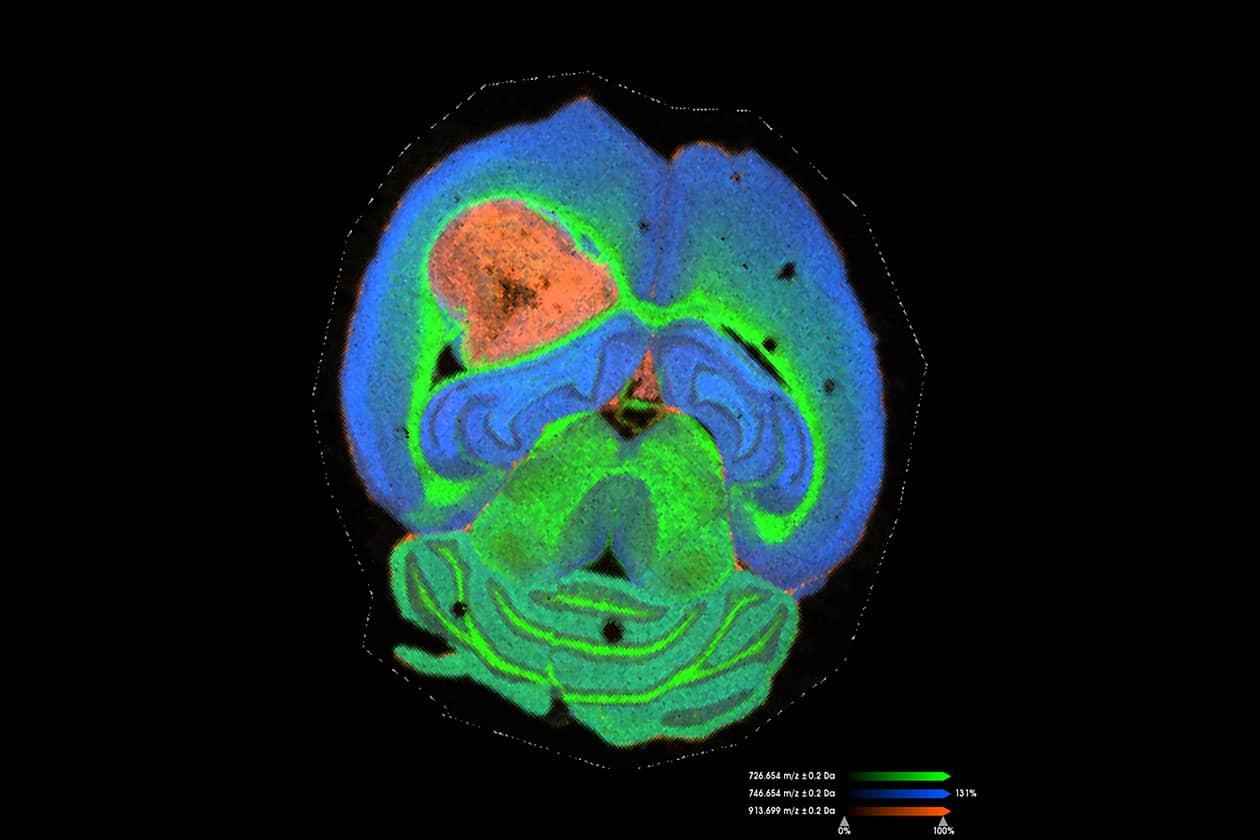
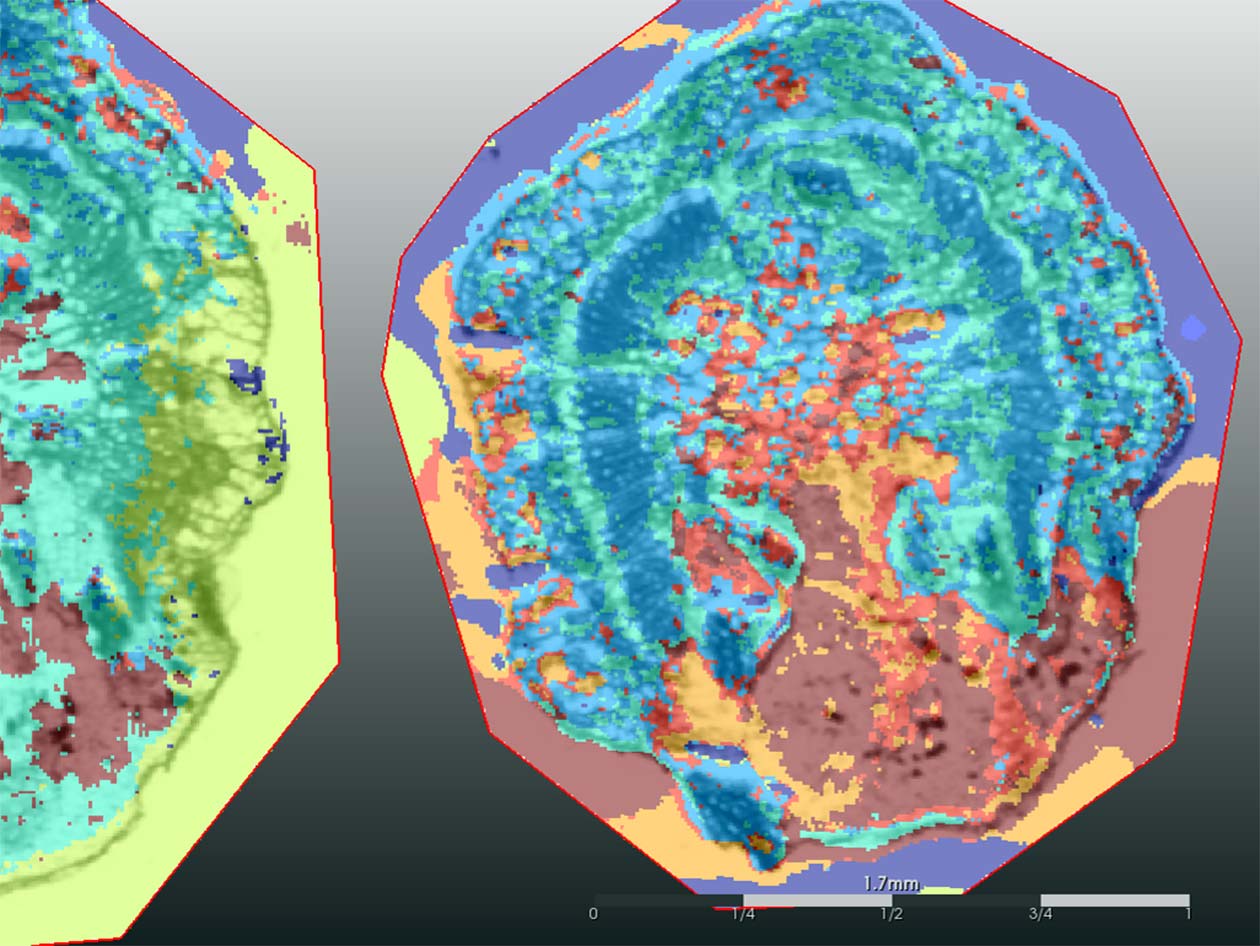
- Imagerie par spectrométrie de masse MALDI et DESI,
- Profilage sur tissus par MALDI, DESI et SpiderMass,
- Analyse protéomique à grande échelle (bottom-up, shotgun, top-down), quantification label-free et analyse de grandes cohortes,
- Analyse de partenaires d’interactions protéine-protéine par cross-linking MS et BioID,
- Protéogénomique et étude du protéome alternatif,
- Analyse de protéomique spatiale à partir de sections histologiques de tissus,
- Analyse de données de protéomique et d’imagerie MS avec des outils d’analyse statistique multivariés et de deep learning,
- Protéomique, lipidomique et glycomique sur des échantillons d’archéologie, de paléontologie et d’héritage culturel,
- Purification et quantification de vésicules extracellulaires à partir de différents fluides,
- Production de modèles cellulaires en 3D à partir de lignées cellulaires ou de cellules primaires (sphéroïdes, organoïdes, bio-impressions),
- Accès à la biobanque de tumoroïdes mammaires humains et canins de la plateforme,
- Quantification absolue de composés dans des tissus par imagerie MS,
- Préparation d’échantillons pour l’imagerie MS (coupe, déparaffinage, lavage, démasquage des antigènes, digestion enzymatique) pour tissus frais congelés, fixés, FFPE et tout matériel biologique tissulaire (organes, biopsies, coupes de tissus, organoïdes, sphéroïdes et vésicules extracellulaires),
- Mise à disposition des instruments de type MALDI pour des analyses de routine, et du compteur de particules NanoSight pour la quantification de vésicules extracellulaires.
Moyens et équipements
Spectrométrie de masse et chromatographie :
- timsTOF fleX (Bruker),
- Orbitrap Eclipse Tribrid (Thermo Scientific) avec ETD, gamme m/z étendue et module FAIMS,
- MALDI-TOF RapiFlex (Bruker),
- ESI Q-Orbitrap Q Exactive Plus, (Thermo Scientific),
- ESI/DESI/REIMS-Q-TOF Xevo G2-XS (Waters),
- MALDI/ESI LTQ-Orbitrap XL (Thermo Scientific),
- MALDI-ESI FT-ICR SolariX XR 9.4 T (Bruker),
- MALDI-TOF/TOF Ultraflex II (Bruker),
- ESI LTQ-Orbitrap Elite (Thermo Scientific),
- ESI LTQ-Orbitrap XL (Thermo Scientific),
- ESI Q-Orbitrap Q Exactive (Thermo Scientific),
- ESI Q-TOF avec séparation par mobilité ionique et mode ETD de type Synapt G2S (Waters),
- NanoLC Evosep One (Evosep),
- HPLC et UFLC Prominence (Shimadzu), UHPLC U3000 (Thermo Scientific),
- NanoUHPLC U3000, NanoLC EASY-nLC II et NanoUHPLC EASY-nLC 1000 (Thermo Scientific), NanoUPLC ACQUITY (Waters).
Préparation d’échantillons :
- Systèmes de micronébulisation ImagePrep (Bruker) et TM-Sprayer M5 (HTX Imaging),
- Système de microdépôt piézoélectrique CHIP1000 microspotter (Shimadzu),
- Système de microextraction Triversa Nanomate (Advion) avec option LESA et LESAplus,
- Système de sublimation manuel (Sigma-Aldrich),
- Cryostat (Leica),
- Système de démasquage des antigènes.
Bioinformatique :
- Serveurs PowerEdge T430 RAID6 32 To (Dell), Power Edge R920 (Dell), Proteome Destroyer 64 Go avec 4 canaux DDR3 RAM et processeur 12 cœurs (Omics Computing), station de calcul GPU Nvidia Tesla K20c (Lenovo),
- Logiciels d’imagerie MS : FlexImaging 4 (Bruker Daltonics) et SCiLS Lab (SCiLS),
- Logiciels de traitement de données : Proteome Discoverer 2.4 (Thermo Scientific) avec XLinkX et ProSightPD 1.1, Scaffold 4.6 et perSPECtives (Proteome Software), Progenesis QI et Progenesis QI pour la protéomique (Nonlinear Dynamics), suites MaxQuant et Perseus, PEAKS 11 et Byonics.
Culture cellulaire :
- Bioimprimante BIO X (Cellink),
- Microscope à fluorescence Eclipse TS2-FL (Nikon),
- Équipements classiques : PSM, incubateurs à CO2 et centrifugeuse.
Purification de vésicules extracellulaires :
- NanoSight NS300 (Malvern),
- Collecteur de fraction automatique (Izon),
- Ultracentrifugeuse (Beckman).




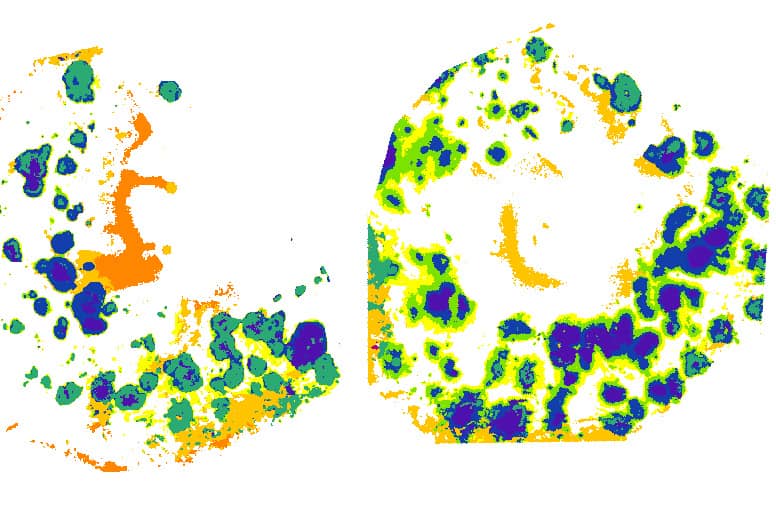

Comment soumettre un projet ?
Pour soumettre une demande à OrganOmics, vous pouvez contacter la plateforme par téléphone ou par mail à l’adresse organomics@univ-lille.fr en décrivant brièvement votre projet. Vous serez par la suite invité à faire une présentation lors d’une rencontre physique ou en visioconférence.
Les projets sont évalués en interne par un comité. Lorsqu’une demande est validée, les moyens techniques sont précisés, un planning prévisionnel est établi et une proposition est adressée au responsable du projet. Pour les demandes plus complexes, une étude préliminaire est réalisée à la charge de la plateforme sur un à trois échantillons afin de vérifier la faisabilité des travaux. La proposition est accompagnée d’un devis, sauf dans le cas où une collaboration scientifique est plus adaptée à la demande.
Cette procédure est valable pour les analyses en imagerie par spectrométrie de masse, les analyses protéomiques et les projets concernant les modèles cellulaires en 3D. Le spectromètre de masse MALDI-TOF/TOF Ultraflex II (Bruker) et le compteur de particules NanoSight (Malvern) mis à disposition par la plateforme peuvent être utilisés en autonomie après formation. L’accès se fait sur réservation d’un créneau et sous condition de disponibilité.
Exemple de projet
Caractérisation de biomarqueurs sanguins pour la sclérodermie
Le projet SCLEROPROT se concentre sur la sclérose systémique (SSc), une maladie auto-immune rare et grave. La SSc est difficile à diagnostiquer et détient le plus mauvais pronostic de toutes les maladies auto-immunes systémiques. L’objectif du projet est d’identifier une signature moléculaire spécifique associée au diagnostic, à l'évolution de la maladie et à la réponse au traitement, par stratégie multiomique, afin d'optimiser la stratification des patients et les traitements dans la pratique clinique et les futurs essais cliniques de phase 3.
Le projet comprend l'analyse de 400 échantillons de plasma provenant de patients atteints de SSc et de témoins sains. Les vésicules extracellulaires ont été isolées du plasma par chromatographie d'exclusion de taille puis analysées, avec les protéines circulantes libres, par LC-MS HR. Ces opérations ont nécessité le savoir-faire des sites MS4Omics et 3DCellOmics de la plateforme OrganOmics. L'objectif final est de développer un score prédictif de la progression de la maladie et de la réponse au traitement en utilisant la signature moléculaire dérivée de l'analyse intégrative des données protéomiques et des données cliniques pertinentes.
Contact
OrganOmics
PRISM, Inserm U1192
Université de Lille, Cité scientifique
Bâtiment SN3, 1er étage
59655 Villeneuve-d’Ascq
Région : Hauts-de-France +33 (0)3 20 43 41 94
+33 (0)3 20 43 41 94
organomics@univ-lille.fr
Site de la plateforme
THÉMATIQUES : Nouvelles thérapies, vectorisation, cellules souches, organoides, Protéomique, Autres
RESPONSABLES SCIENTIFIQUES :
Isabelle Fournier, Marie Duhamel
RESPONSABLES TECHNIQUES :
Fabrice Bray, Antonella Raffo-Romero
TUTELLES : Centre Oscar Lambret, CHU de Lille, CNRS, Inserm, Université de Lille
INFRASTRUCTURES NATIONALES : ProFI
LABELLISATION IBiSA : 2008
MOTS CLÉS : Imagerie par spectrométrie de masse, Protéomique spatiale, Shotgun, Bottom-up, Middle-down, Top-down, Cross-linking MS, Marquage de proximité, BioID, TurboID, Bioprinting, Sphéroïdes, Tumoroïdes, Organoïdes, Vésicules extracellulaires, Analyses à large échelle, CAR-macrophages, MALDI-IHC, Paléoprotéomique, Héritage culturelle, Foodomics
Fiche mise à jour en 2023










