IBPC Plateformes
Caractérisation des macromolécules biologiques, de leurs interactions et de leurs dynamiques, avec contrôle de la qualité des échantillons complexes et visualisation à différentes échelles.

IBPC Plateformes
Caractérisation des macromolécules biologiques, de leurs interactions et de leurs dynamiques, avec contrôle de la qualité des échantillons complexes et visualisation à différentes échelles.
IBPC Plateformes
L’institut de biologie physico-chimique (IBPC) a pour vocation de promouvoir des recherches dans tous les domaines de la biologie. Les bases structurales, génétiques et physicochimiques du vivant y sont étudiées à leurs différents niveaux d’intégration, de la molécule à la cellule. Les plateformes de l’IBPC ont été montées et développées grâce à l’EquipEx CACSICE et au LabEx DYNAMO obtenus en 2012. Quatre d’entre elles sont ouvertes à la communauté scientifique : la plateforme de RMN biomoléculaire, la plateforme de protéomique, la plateforme de cristallographie et biophysique et la plateforme de visualisation. Les plateformes de l’IBPC s’appuient sur les expertises de ses unités de recherche.
Expertises et services
RMN liquide et solide :
- Mise à disposition de spectromètres RMN pour une utilisation en autonomie,
- Réalisation de spectres 1D 1H, 2D 1H-15N...,
- Contrôle qualité des échantillons produits,
- Étude de protéines et d’ARNs,
- Analyse d’interactions protéine-ligand, ARN-ligand et protéine-ARN,
- Exploration de paysages énergétiques et de dynamiques conformationnelles.
Protéomique :
- Analyse protéomique à partir de protéines purifiées ou d’échantillons protéiques complexes,
- Identification de protéines,
- Contrôle qualité des protéines recombinantes,
- Quantification relative de protéomes,
- Analyse quantitative label-free,
- Caractérisation de modifications post-traductionnelles ou chimiques,
- Préparation d’échantillons.
Cristallographie et biophysique :
- Mise à disposition d'équipements pour une utilisation en autonomie,
- Cristallisation de protéines membranaires en phase cubique lipidique (LCP),
- Criblage en gouttes de nanovolumes sur microplaques pour la recherche de conditions de cristallisation et mise à disposition des clichés des gouttes obtenues jour après jour,
- Mesure d’affinités par microcalorimétrie ITC et détermination des paramètres thermodynamiques d’interaction,
- Mesure de la masse de macromolécules et de leurs complexes par SEC-MALS.
Visualisation :
- Visualisation d’images de microscopie à haute résolution,
- Visualisation de la structure de macromolécules et des trajectoires de simulation en mode stéréoscopique et en réalité virtuelle,
- Aide à la résolution de structures cristallographiques ou cryo-EM,
- Visualisation en groupes d’environ 10 personnes.
Moyens et équipements
RMN liquide et solide :
- Spectromètre Bruker Avance III HD 700 MHz avec console TwoBay,
- Cryo-sonde liquide TCI 5 mm : TCI 700 S3 H&F-C/N-D-05 Z,
- Sonde solide 3,2 mm : PH MAS 700 S3 1H/13C/79Br/31P/15N,
- Sonde solide 4 mm : PH MAS 700 S4 BB-1H,
- Sonde liquide TXI 5 mm : PA TXI 700 S3 1H-13C/15N-D-05 Z,
- Sonde liquide BBO 10 mm : PA BBO 700 S3 BB-1H-D-10 Z.
Protéomique :
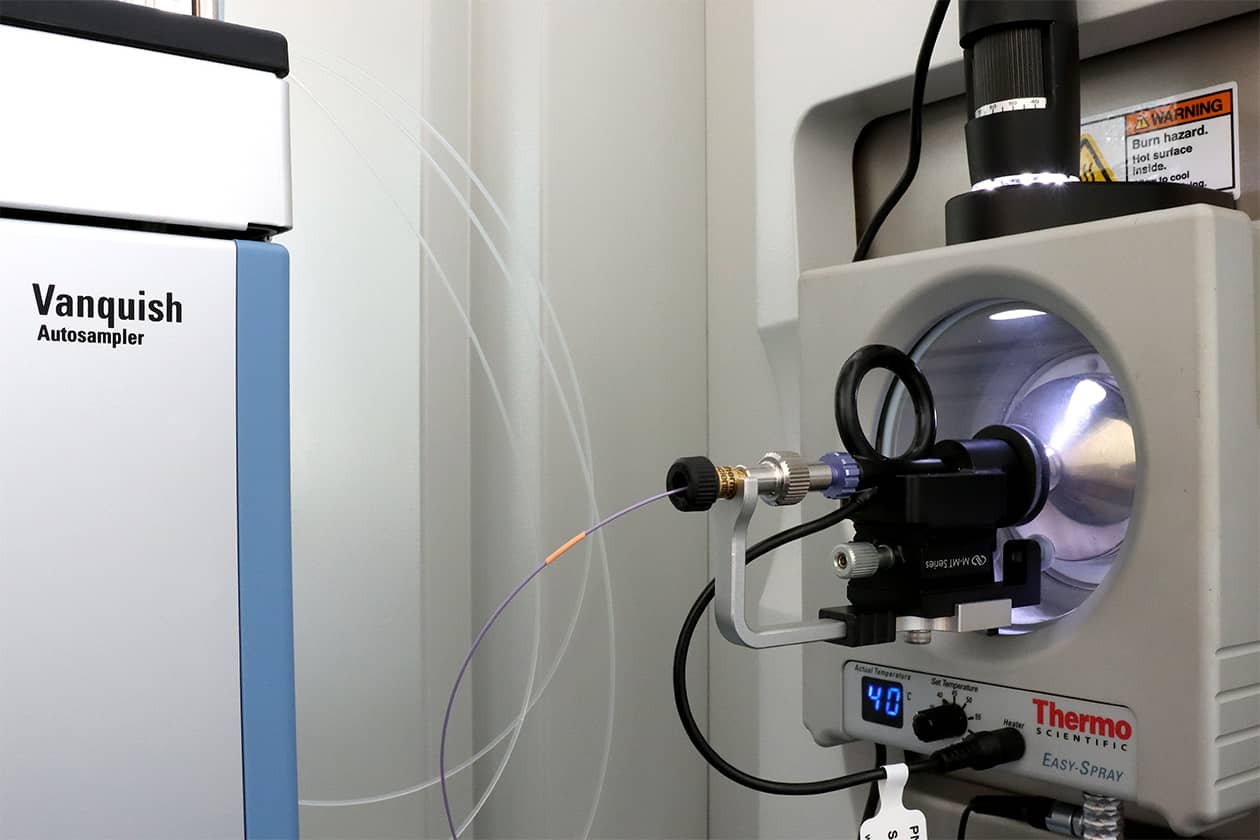
- Spectromètre de masse Q Exactive Plus Thermo Scientific relié à une UPLC Vanquish Neo.
Cristallographie et biophysique :
- Robots pipeteurs Mosquito LCP STP Labtech 96 puits à 4°C et 18°C,
- Robot d’optimisation Dragonfly STP Labtech 96 puits,
- Robots de visualisation de cristaux Imascope et Explora Nova à 4°C et 18°C,
- Serveur de stockage pour les images de cristaux avec interface d’observation Imascope,
- Microscopes binoculaires Zeiss et Nikon,
- Microcalorimètre PEAQ-ITC Malvern Panalytical.
Visualisation :
- Mur d’écrans stéréoscopiques de grande taille (4,4 m de largeur x 1,8 m de hauteur) et de haute résolution (7 680 x 3 240 px),
- Système de réalité virtuelle HTC Vive.





Comment soumettre un projet ?
Pour soumettre un projet à IBPC Plateformes, vous pouvez contacter directement les responsables des équipements concernées : RMN liquide et solide, protéomique, cristallographie, biophysique ou visualisation. Une réponse vous sera apportée dans les meilleurs délais.
Exemple de projet
Caractérisation structurale de l'interface de dimérisation de l’enzyme ADAR1
Enzyme clé de l'édition des ARN, la protéine ADAR1 catalyse la conversion des adénosines en inosines au sein des ARN double brin. Cette activité joue un rôle essentiel dans la reconnaissance et la distinction entre les ARNs endogènes et exogènes. Dans le cadre d'un projet, dont l’objectif était de caractériser l’interface de dimérisation de l’enzyme ADAR1, plusieurs plateformes de l’IBPC ont apporté leur expertise.
La plateforme de cristallographie a mis à disposition ses robots de cristallisation pour effectuer des criblages larges, ce qui a permis d’identifier les premières conditions de cristallisation du troisième domaine de liaison aux ARNs double brin d’ADAR1, appelé ADAR1-dsRBD3, seul ou en complexe avec un ARN double brin. La plateforme a apporté son savoir-faire pour l’optimisation des cristaux obtenus afin d’améliorer leur qualité de diffraction. Ce travail a permis de résoudre la structure d’ADAR1-dsRBD3, à la fois libre et lié à une double hélice d’ARN. Une analyse structurale a révélé que ce domaine permettait la dimérisation de l’enzyme au travers d’une interface étendue, impliquant notamment des interactions entre feuillets bêta.
En parallèle, la plateforme de RMN biomoléculaire a été mobilisée pour étudier le repliement de différents mutants d’ADAR1-dsRBD3 conçus pour perturber cette interface de dimérisation et rendre le domaine monomérique. Les expériences menées sur ces variants, porteurs de mutations ponctuelles, ont montré que les mutations empêchaient effectivement la dimérisation, sans altérer la solubilité ni compromettre le repliement intrinsèque du domaine. Les données structurales obtenues grâce à IBPC Plateformes ont été déterminantes pour comprendre le rôle fonctionnel de la dimérisation d’ADAR1 dans un contexte cellulaire.
Pour en savoir plus : Mboukou A. et al. (2024). Dimerization of ADAR1 modulates site-specificity of RNA editing. Nature Communications, 15 (1):10051.
Contact
IBPC Plateformes
13 rue Pierre et Marie Curie
75005 Paris
Région : Île-de-France +33 (0)1 58 41 51 00
+33 (0)1 58 41 51 00
tisne@ibpc.fr
Site de la plateforme
THÉMATIQUES : Biologie structurale, biophysique, biochimie, Protéomique
RESPONSABLES SCIENTIFIQUES :
Carine Tisné
RESPONSABLES TECHNIQUES :
Christel Le Bon
TUTELLES : CNRS
INFRASTRUCTURES NATIONALES : ProFI
LABELLISATION IBiSA : 2024
MOTS CLÉS : RMN solide-liquide, Assemblages supramoléculaires, Spectrométrie de masse, Q Exactive Plus, LC-MS/MS, Protéomique, Quantification, Visualisation, Stéréoscopie, Réalité virtuelle, Mur d’images, Protéine, ARN, Cristallisation, Phase cubique lipidique, LCP, Structure, Microcalorimétrie, Interactions
Fiche mise à jour en 2025










