Plateforme d’exploration du métabolisme (PFEM)
Développement d’outils et de méthodes en métabolomique, fluxomique et protéomique pour des applications en nutrition, santé, environnement et qualité des produits alimentaires.

Plateforme d’exploration du métabolisme (PFEM)
Développement d’outils et de méthodes en métabolomique, fluxomique et protéomique pour des applications en nutrition, santé, environnement et qualité des produits alimentaires.
Plateforme d’exploration du métabolisme (PFEM)
La plateforme PFEM a pour objectif de mettre à disposition de la communauté scientifique des concepts, méthodes et outils dédiés à l’exploration du métabolisme. Elle constitue un outil commun pour l'étude du métabolome environné, c'est-à-dire enrichi d'une capacité d'analyse en protéomique. Elle propose aux équipes de recherche des technologies d'analyse globale fondées sur la spectrométrie de masse et la RMN, en garantissant une utilisation optimale des instruments et un traitement approprié des données produites. La plateforme possède également de fortes compétences en statistiques et bioinformatique. Enfin, elle est l’un des quatre membres fondateurs de l’infrastructure nationale d’excellence MetaboHUB.
Expertises et services
- Métabolomique non ciblée : détermination de profils métaboliques et identification de biomarqueurs sur biofluides et tissus,
- Quantification de petites molécules dans les différents compartiments d'organismes,
- Fluxomique : mesure d'enrichissement isotopique pour le calcul de débits métaboliques,
- Protéomique : identification, caractérisation et quantification label-free de protéines et de peptides, protéomique ciblée, imagerie sur coupes histologiques.
Moyens et équipements
- Métabolomique non ciblée : UPLC-QTOF Impact HD II Bruker (ESI et APCI), UPLC-QTOF timsTOF Bruker (ESI), GC-QTOF 7200 Agilent (EI et CI), RMN 500 MHz Ultrashield Plus Avance III Bruker (sonde Prodigy TCI 5 mm),
- Identification structurale : UPLC-LTQ Orbitrap Velos Thermo Scientific (ESI et APCI), source Triversa Nanomate Advion,
- Métabolomique ciblée : 5500 QTRAP AB Sciex (ESI et APCI) couplé à une chaîne SFC/UPLC 1260 Infinity Agilent, GC-MS/MS Quattro Micro Waters (EI et CI), LC-MS/MS Quattro Micro Waters (ESI et APCI), 2 GC-MS 7890B Agilent (EI et CI),
- Fluxomique : 2 GC-MS 7890B Agilent (EI et CI), GC-C-IRMS Isoprime GV Instruments (EI),
- Protéomique : MALDI-TOF/TOF Autoflex Speed Bruker équipé d’un module d’imagerie, Q Exactive HF-X Thermo Scientific (ESI) couplé à une nano HPLC Ultimate RSLC 3000, LTQ Orbitrap Velos Thermo Scientific (ESI) couplé à une micro HPLC Ultimate RSLC 3000,
- Préparation d’échantillons : Freedom EVO 200 Tecan et SamplePro Tube Bruker,





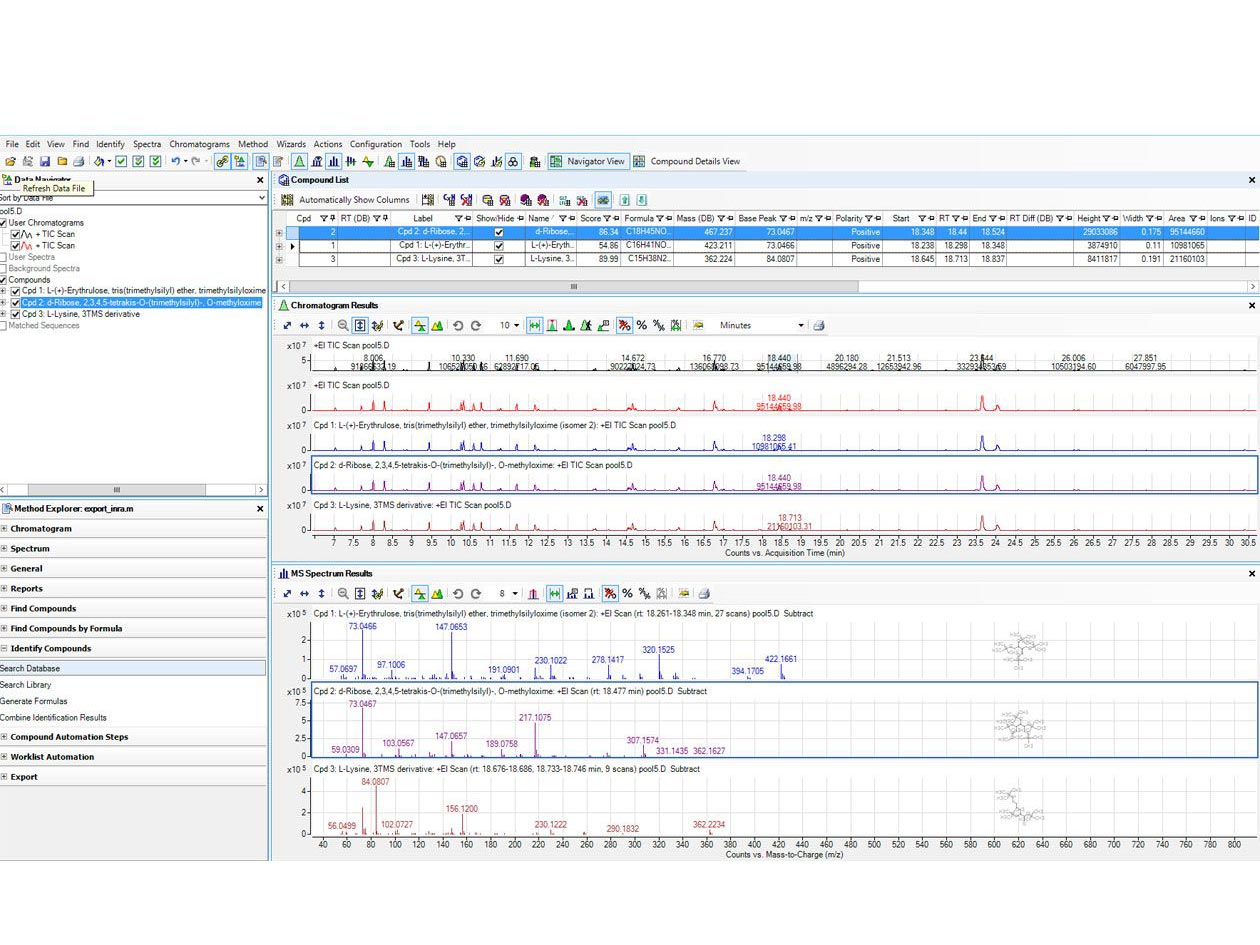

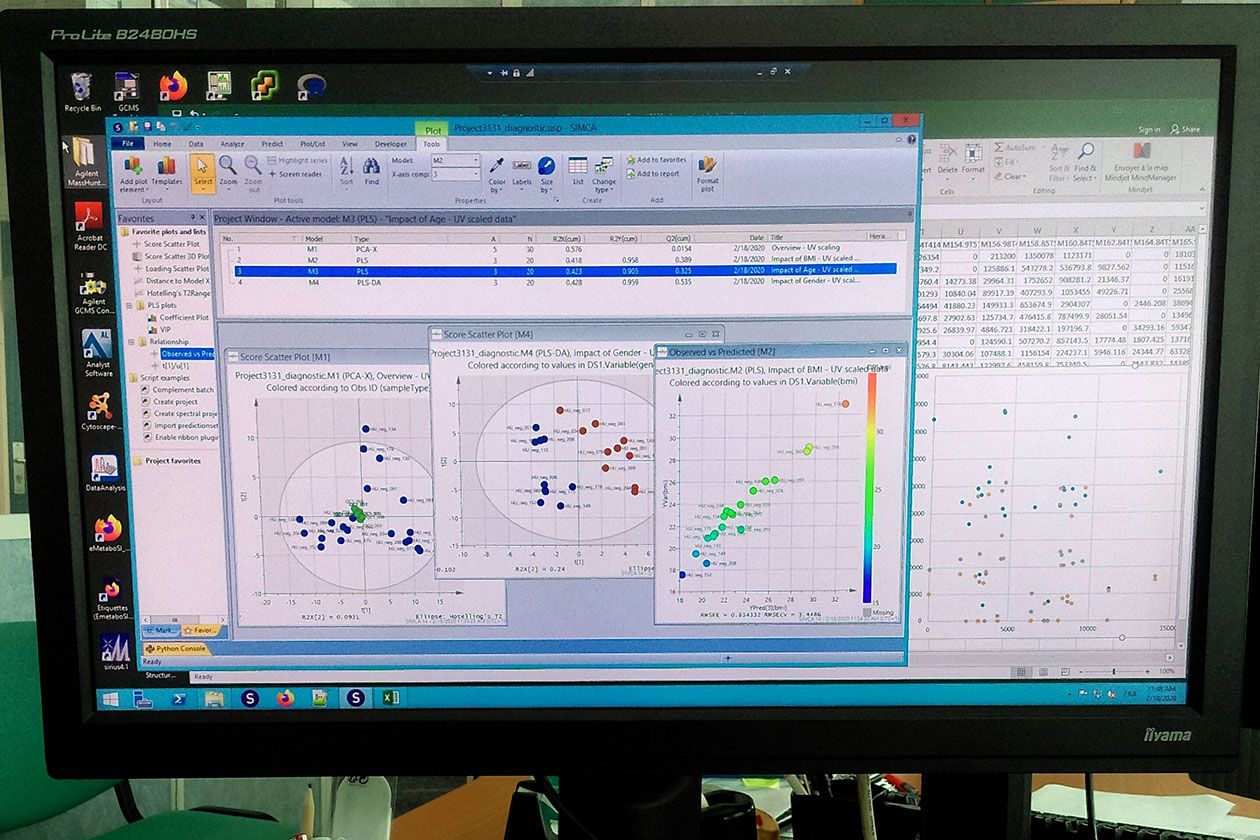
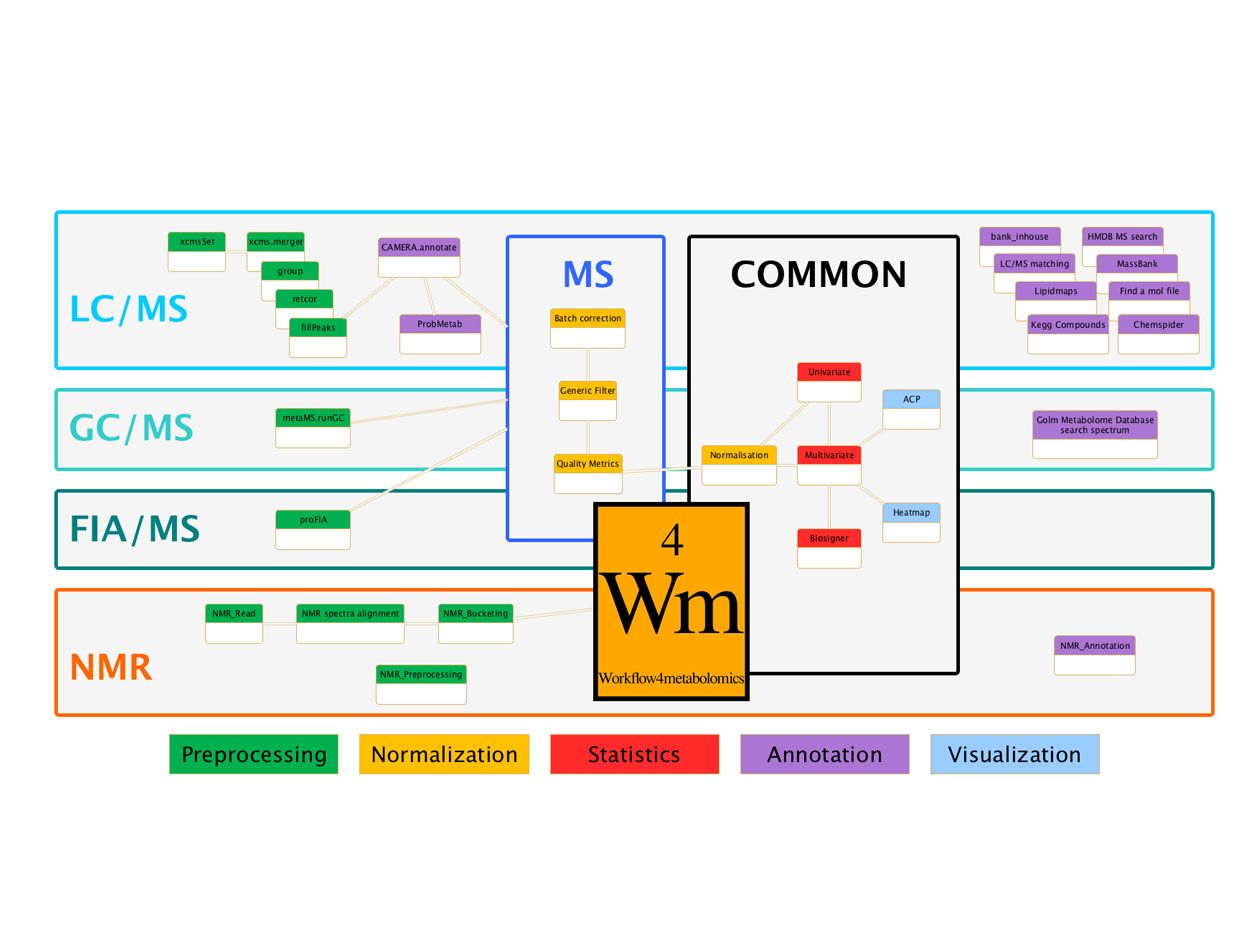

Comment soumettre un projet ?
Pour accéder à un service ou proposer une collaboration en métabolomique et fluxomique à la plateforme PFEM, le portail MAMA est à votre disposition sur le site de l’infrastructure MetaboHUB. Il permet d’enregistrer les demandes, d’échanger autour des besoins, de réaliser des études de faisabilité et de proposer des solutions adaptées.
Les demandes de collaboration en protéomique se font par prise de contact directe avec les responsables de la composante impliquée. Cet échange permet aussi de cerner les besoins, de réaliser des études de faisabilité et de proposer des services adaptés.
Exemple de projet
Utiliser la métabolomique pour décrypter les relations nutrition-santé
Dans le cadre de recherches sur la prévention des désordres métaboliques liés au vieillissement, le groupe Mapping dirigé par Blandine Comte a coordonné un projet visant à identifier des biomarqueurs prédictifs du syndrome métabolique (SMet) chez l’Homme.
Mené en étroite collaboration avec la plateforme PFEM, le projet a consisté en une étude cas-témoin réalisée au sein de la cohorte Gazel. Des échantillons de sérum prélevés cinq ans avant le diagnostic du syndrome ont été analysés en LC/MS par métabolomique non ciblée. Des méthodes de fouille de données ont été développées pour sélectionner les meilleurs candidats biomarqueurs. Les données métabolomiques ont ensuite été intégrées aux paramètres phénotypiques afin de construire des modèles multidimensionnels.
Les résultats ont montré qu’un profil métabolique réduit permettait de révéler des différences phénotypiques subtiles 5 ans avant l’apparition du SMet. L’intégration aux données cliniques des marqueurs métabolomiques obtenus a permis d’améliorer les capacités prédictives des modèles. Ce travail démontre l’intérêt d’une telle approche de phénotypage pour la stratification de patients.
Pour en savoir plus : Pujos-Guillot E. et al. (2017). Systems metabolomics for prediction of metabolic syndrome. Journal of Proteome Research, 16(6):2262-2272.
Contact
PFEM
INRAE, Centre de recherche de Clermont-Ferrand - Theix
63122 Saint-Genès-Champanelle
Région : Auvergne-Rhône-Alpes +33 (0)4 73 62 41 55
+33 (0)4 73 62 41 55
contact-pfem@inrae.fr
Site de la plateforme
THÉMATIQUES : Bioinformatique, bases de données, Métabolomique, exposome, Protéomique
RESPONSABLES SCIENTIFIQUES :
Estelle Pujos-Guillot
RESPONSABLES TECHNIQUES :
Stéphanie Durand
RESPONSABLES QUALITÉ :
Josie Colombet
TUTELLES : INRAE, Université Clermont Auvergne
INFRASTRUCTURES NATIONALES : MetaboHUB
LABELLISATION IBiSA : 2010
MOTS CLÉS : Métabolomique, Fluxomique, Protéomique, Bioinformatique, Statistiques, LC-HRMS, GC-HRMS, RMN, IRMS, Métabolisme, Biomarqueurs
Fiche mise à jour en 2021












