Plateforme histopathologie haute précision (H2P2)
Une expertise et un savoir-faire de haut niveau en histopathologie pour la communauté scientifique académique et privée.
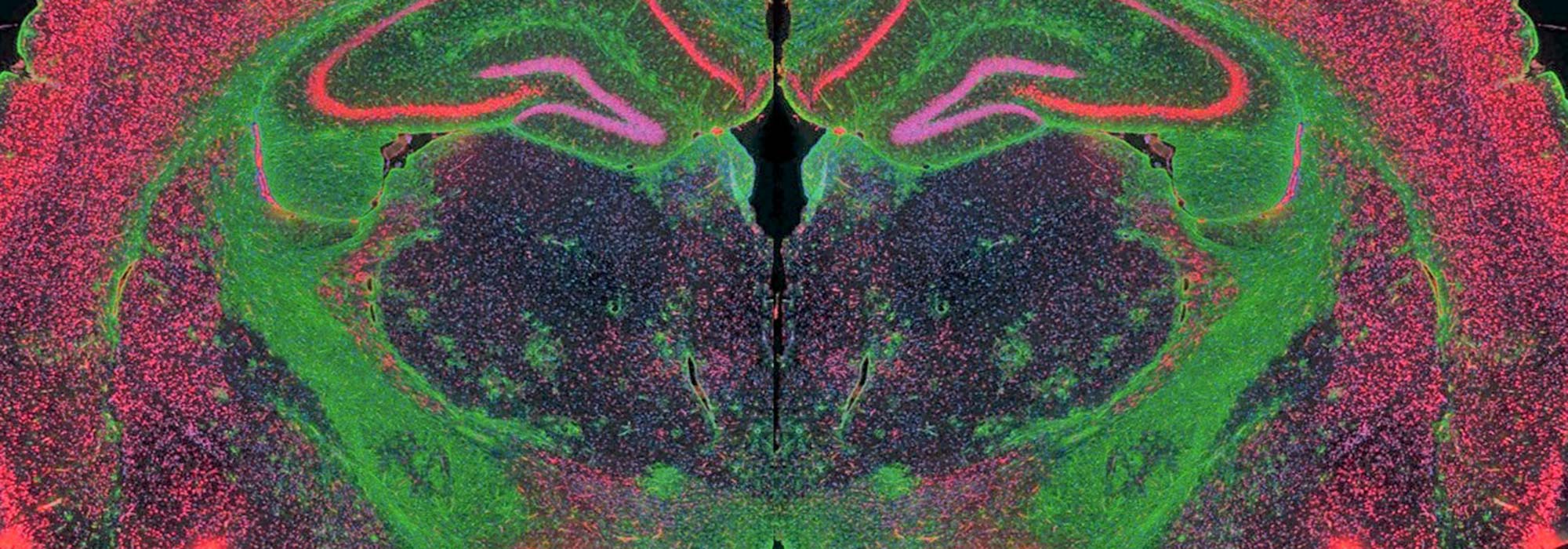
Plateforme histopathologie haute précision (H2P2)
Une expertise et un savoir-faire de haut niveau en histopathologie pour la communauté scientifique académique et privée.
Plateforme histopathologie haute précision (H2P2)
La plateforme H2P2 propose une expertise et un savoir-faire en histopathologie dans des domaines tels que la santé, l’agroalimentaire, la cosmétologie et l’environnement. En plus de son offre de service couvrant les besoins de base en histologie, elle a développé des compétences sur des technologies de pointe. Son expertise en immunomarquage simple et multiplex permet de mettre en évidence jusqu’à 6 protéines différentes par lame, en fluorescence ou chromogénie. H2P2 est de plus la référence européenne pour la technologie hyperplex Cell DIVE avec une détection de 30 cibles par lame. La plateforme visualise ses lames à l’aide d’un microscope épifluorescent et de scanners de lames. Elle réalise également des tissue microarrays (TMA) automatisés permettant de regrouper des centaines de tissus sur une même lame. Elle possède par ailleurs une expertise en microdissection par capture laser, transparisation d’échantillons biologiques et imagerie par microspectroscopie Raman, CARS, SRS et SHG.
Après formation, H2P2 met différents équipements à disposition de la communauté scientifique. La plateforme collabore également sur les projets de recherche de ses utilisateurs. Elle apporte son expertise concernant l’ensemble de ses activités, optimise les marquages immunohistochimiques en simplex ou multiplex et met en évidence des ARNs d’intérêt par hybridation in situ sur des coupes de tissus et TMA réalisés en interne. La microdissection par capture laser permet de sélectionner les cellules d’intérêt sur des coupes tissulaires, puis d’analyser leur transcriptome par microarray, NGS ou RT-qPCR. La microspectroscopie Raman permet quant à elle d’évaluer les modulations phénotypiques des cellules par la mise en évidence de signatures spectrales.
Expertises et services
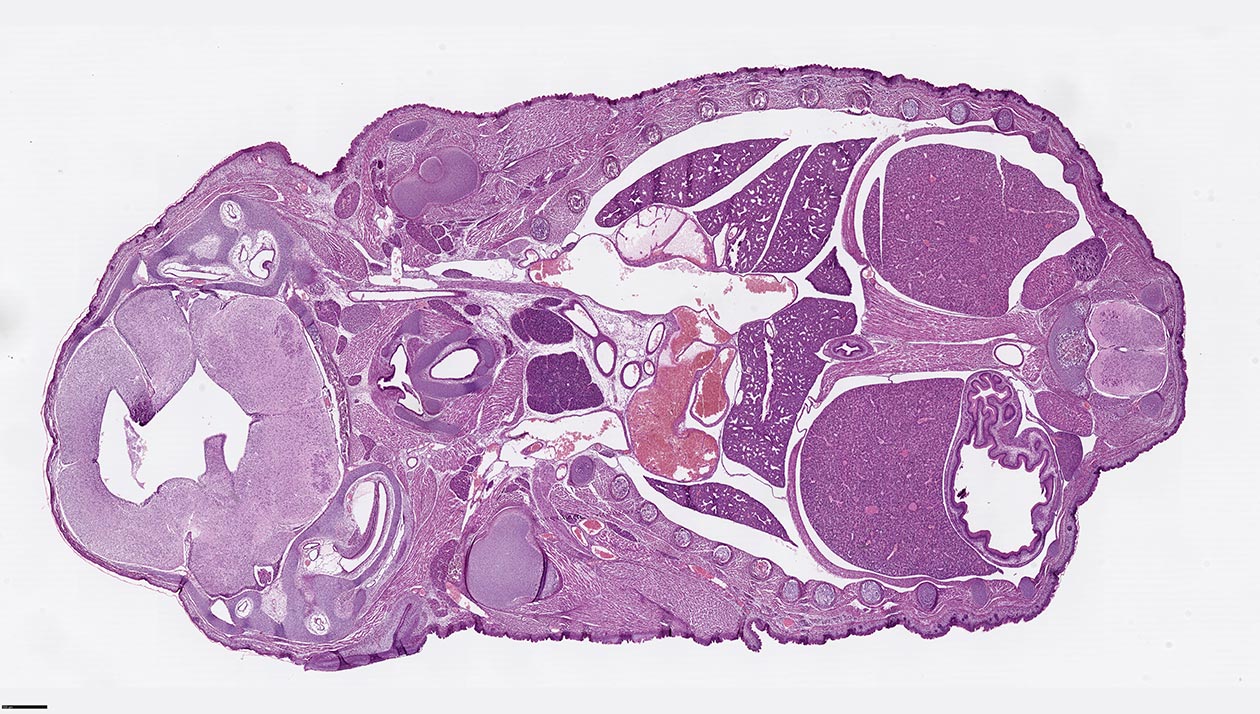
- Histologie classique : fixation de tissus humains, animaux et végétaux, imprégnation en paraffine, réalisation de coupes fines et de colorations histologiques,
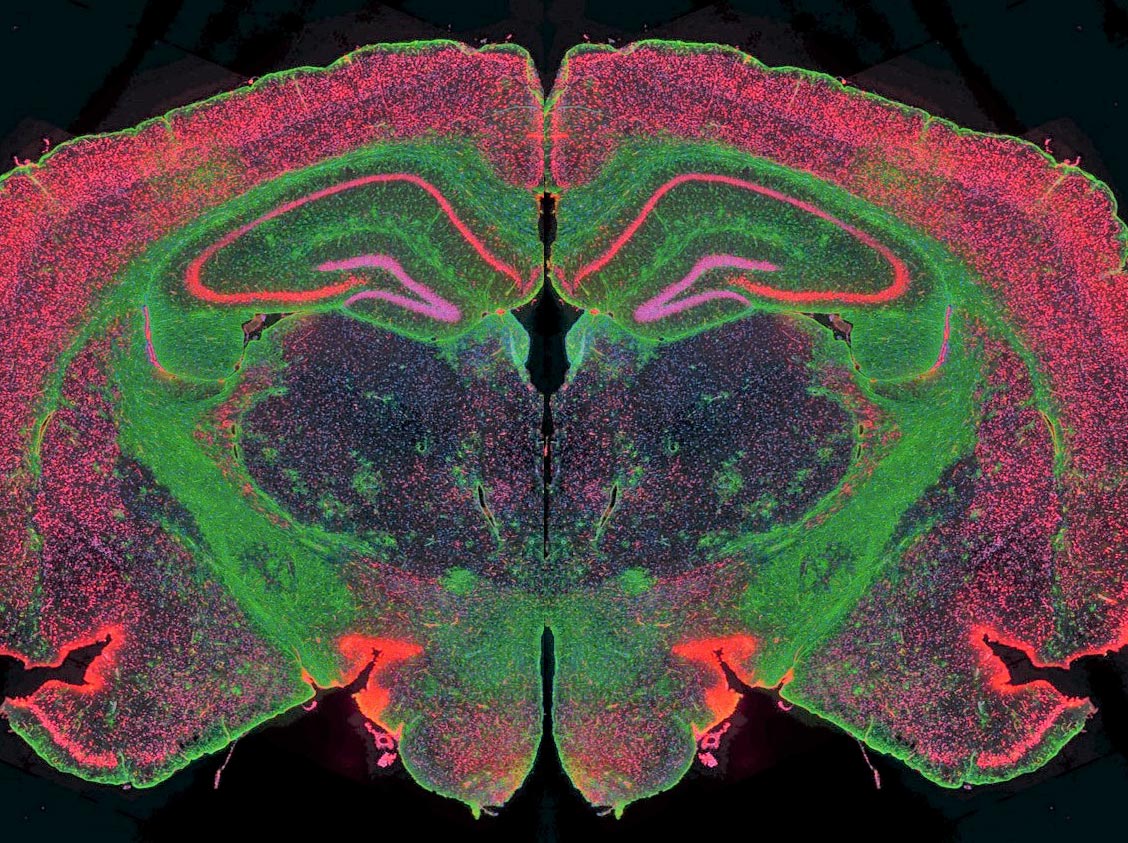
- Immunohistochimie multiplexe : immunomarquage simple et multiplex avec près de 1 500 anticorps au catalogue permettant de mettre en évidence jusqu’à 6 protéines par lame, en fluorescence ou chromogénie , marquage immunohistochimique de cellules, tissus et organes,
- Immunohistochimie hyperplexe : détection de 30 cibles sur une même lame grâce à la technologie Cell DIVE et en partenariat avec Leica Microsystems,
- Réalisation d’hybridations in situ,
- Réalisation de tissue microarrays (TMA) automatisés permettant de regrouper des centaines de tissus sur une même lame,
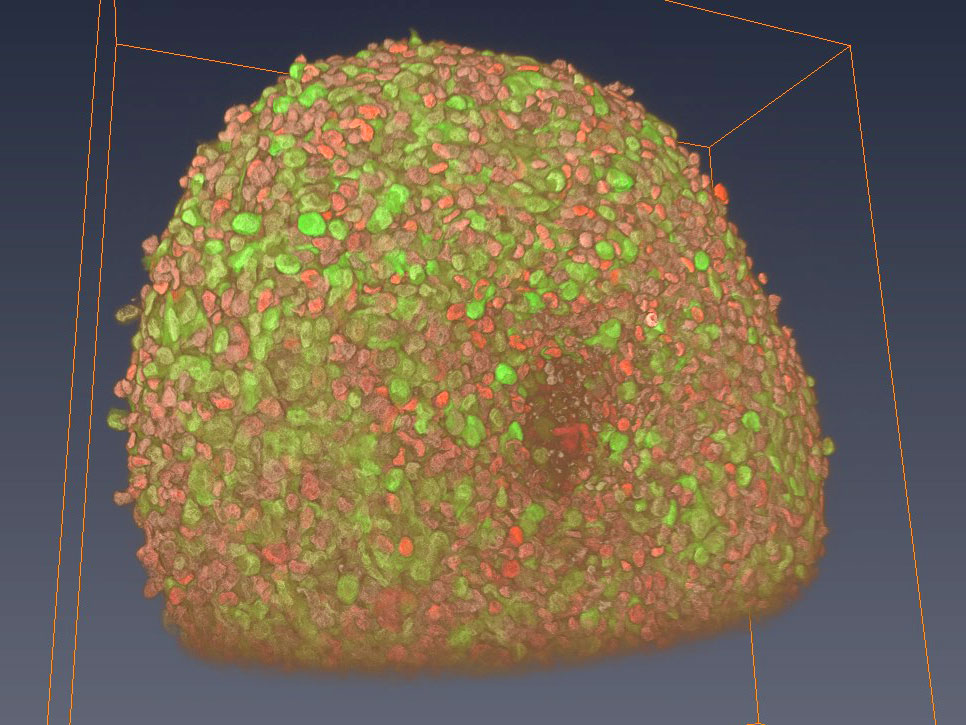
- Transparisation et clarification d’échantillons pour l’imagerie de fluorescence (protocole CUBIC), avec ou sans immunodétection in toto, et reconstitution 3D,
- Numérisation de lames virtuelles à l’aide d’un microscope épifluorescent et de scanners à fond clair et fluorescence,
- Microdissection par capture laser à partir de coupes histologiques d’origine humaine, murine, piscicole et végétale, avec capture individuelle de cellules pour l’analyse du transcriptome,
- Imagerie de lipides, protéines et métabolites secondaires sans marquage, imagerie par microspectroscopie Raman, CARS, SRS et SHG,
- Automatisation de l’analyse d’images en fond clair et fluorescence avec développement de scripts, pré-traitement des données et statistiques avec Python et R,
- Mise à disposition d’équipements d’histologie après formation.
Moyens et équipements
- Automate de déshydratation Excelsior AS,
- Automate d'inclusion HistoStar,
- Automate de congélation rapide SnapFrost,
- 5 microtomes et 2 cryostats pour les coupes fines,
- Vibratome pour la coupe de tissus frais,
- Secteur de culture cellulaire pour la mise en place de cultures organotypiques à partir de tranches tissulaires réalisées avec le vibratome,
- Automate MiniCore 3 pour la réalisation de tissue microarrays (TMA),
- Automate de coloration Leica ST5020,
- Monteuse de lamelles Leica CV5030,
- 2 automates d’immunohistochimie et d'hybridation in situ Roche XT et Ultra pour les marquages simplex et multiplex,
- Automate d’immunohistochimie pour le marquage de tissus entiers (embryons...),
- Imageur multiplex Cell DIVE pour la cartographie spatiale des biomarqueurs et l’étude du microenvironnement tissulaire,
- Microdissecteur par capture laser Arcturus Veritas,
- Microspectromètre Raman DXR2xi,
- Scanner de lames Hamamatsu NanoZoomer RS et S60,
- Scanner confocal 3D Histech à fond clair et fluorescence,
- Microscope à fluorescence Nikon NI-E,
- Microscope BonXplorer pour l’imagerie simultanée selon différentes modalités, notamment l’imagerie SRS des liaisons CH2 et CH3, l’imagerie CARS, la fluorescence 2P et l’imagerie SHG,
- Logiciels d’analyse d’images QuPath, HALO, Fiji et Aivia Community,
- Logiciel de reconstruction 3D Napari.
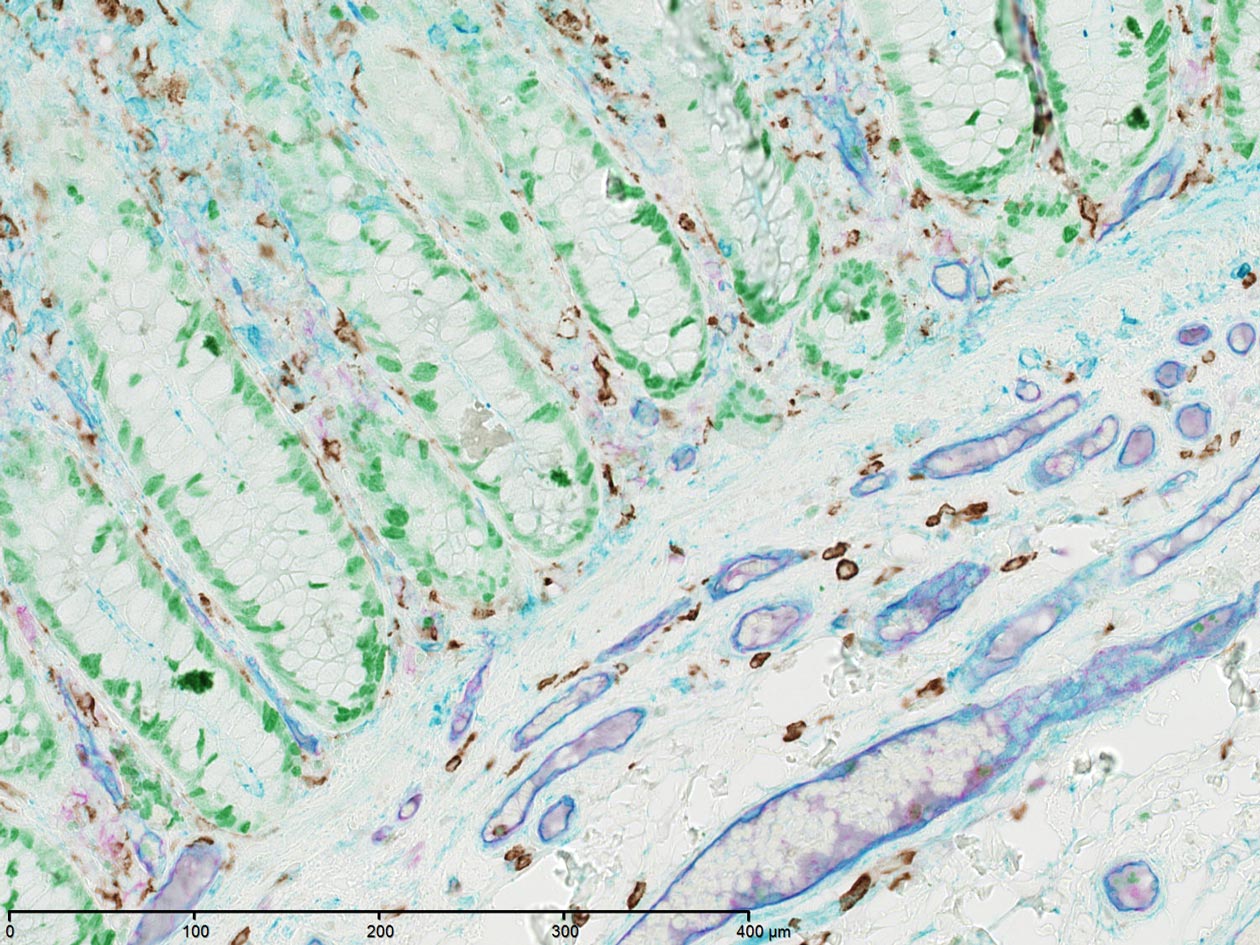
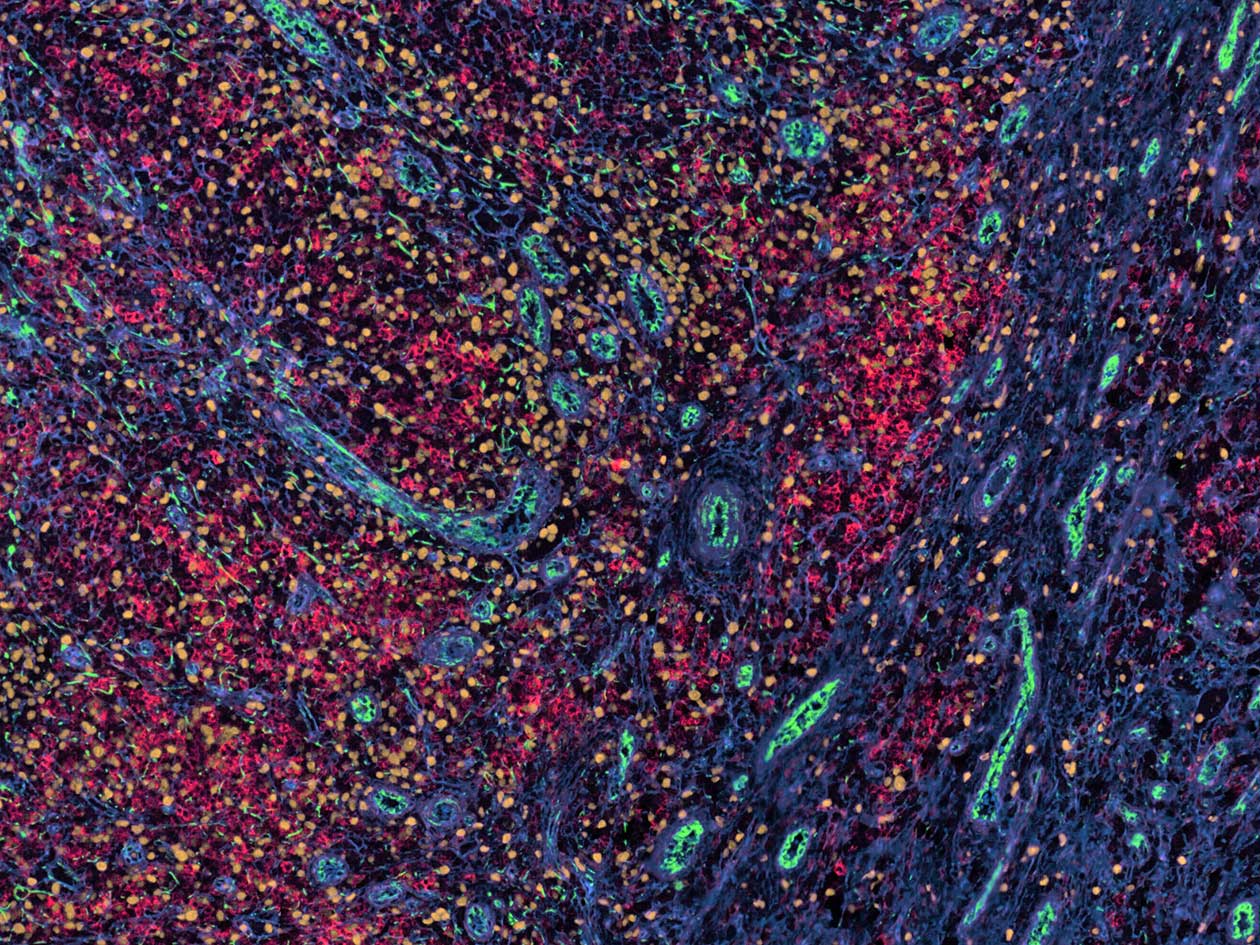




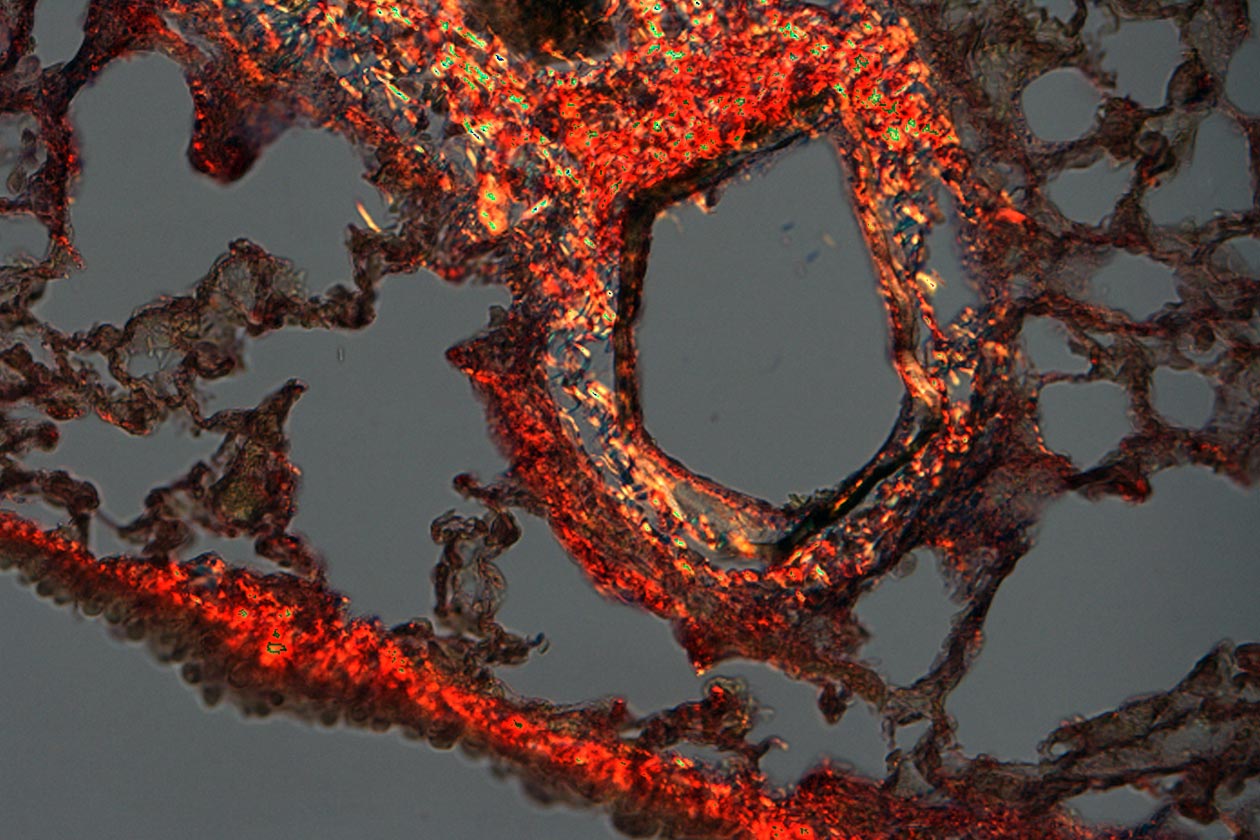

Comment soumettre un projet ?
Pour soumettre un projet à la plateforme, envoyez un mail à l’adresse H2P2@univ-rennes.fr en décrivant brièvement votre projet et le type d’échantillons à analyser. L’équipe d’H2P2 vous recontactera pour prendre un rendez-vous. Elle étudiera avec vous la meilleure stratégie à mettre en œuvre et vous précisera les services qu'elle peut vous offrir, ainsi que les coûts et les délais de réalisation associés.
Exemple de projet
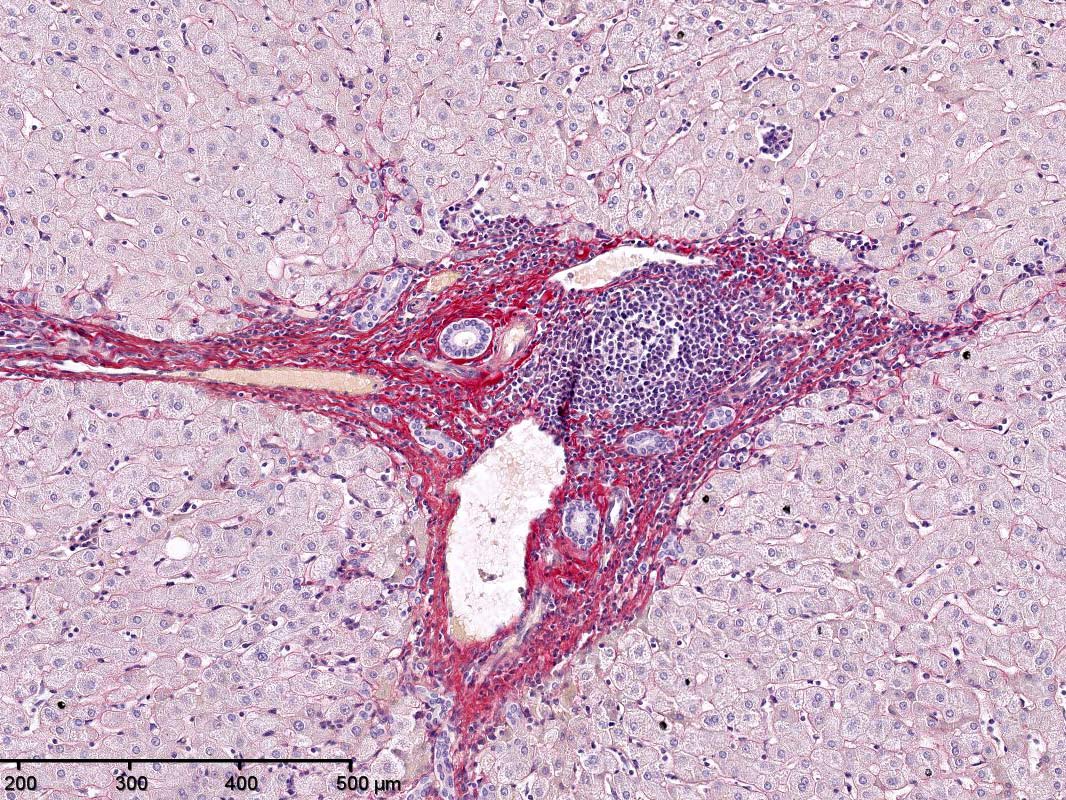
Visualisation du matrisome des nids de fibrose du carcinome hépatocellulaire
La plateforme H2P2 a participé à la détection de 10 protéines de la matrice extracellulaire et de 3 protéines dites « repères topographiques » par immunomarquage multiplex au sein des nids de fibrose de haut grade de 3 carcinomes hépatocellulaires (CHC) humains. La détection des 13 protéines a été réalisée à partir d'une même cryo-coupe.
Pour en savoir plus : Desert R. et al. (2023). Hepatocellular carcinomas, exhibiting intratumor fibrosis, express cancer-specific extracellular matrix remodeling and WNT/TGFB signatures, associated with poor outcome. Hepatology, 78(3):741-757.
Contact
H2P2
Université de Rennes
2 avenue du Pr Léon Bernard
35043 Rennes
Région : Bretagne +33 (0)2 23 23 47 95
+33 (0)2 23 23 47 95
H2P2@univ-rennes.fr
Site de la plateforme
THÉMATIQUES : Histologie, anatomopathologie
RESPONSABLES SCIENTIFIQUES :
Marie-Dominique Gallibert, Bruno Turlin, Orlando Musso
RESPONSABLES TECHNIQUES :
Anthony Sébillot
RESPONSABLES QUALITÉ :
Pascale Bellaud, Anthony Sébillot
TUTELLES : CNRS, Inserm, Université de Rennes
INFRASTRUCTURES NATIONALES : France-BioImaging
LABELLISATION IBiSA : 2014
MOTS CLÉS : Histopathologie, Colorations tissulaires, Immunofluorescence, Immunohistochimie, Simplex, Multiplex, Hyperplex, Cell DIVE, Hybridation in situ, Tissue microarrays, Microdissection par capture laser, Transparisation d’échantillons biologiques, Culture organotypique, Numérisation de lames, Microspectrométrie RAMAN, CARS, SRS, p-SHG, Analyse d’images, Reconstruction 3D, Biogenouest
Fiche mise à jour en 2024










