Plateforme intégrée de biophysique et de biologie structurale (PIBBS)
Des outils de biologie et biophysique pour la caractérisation structurale de macromolécules biologiques, de leur dynamique et associations.

Plateforme intégrée de biophysique et de biologie structurale (PIBBS)
Des outils de biologie et biophysique pour la caractérisation structurale de macromolécules biologiques, de leur dynamique et associations.
Plateforme intégrée de biophysique et de biologie structurale (PIBBS)
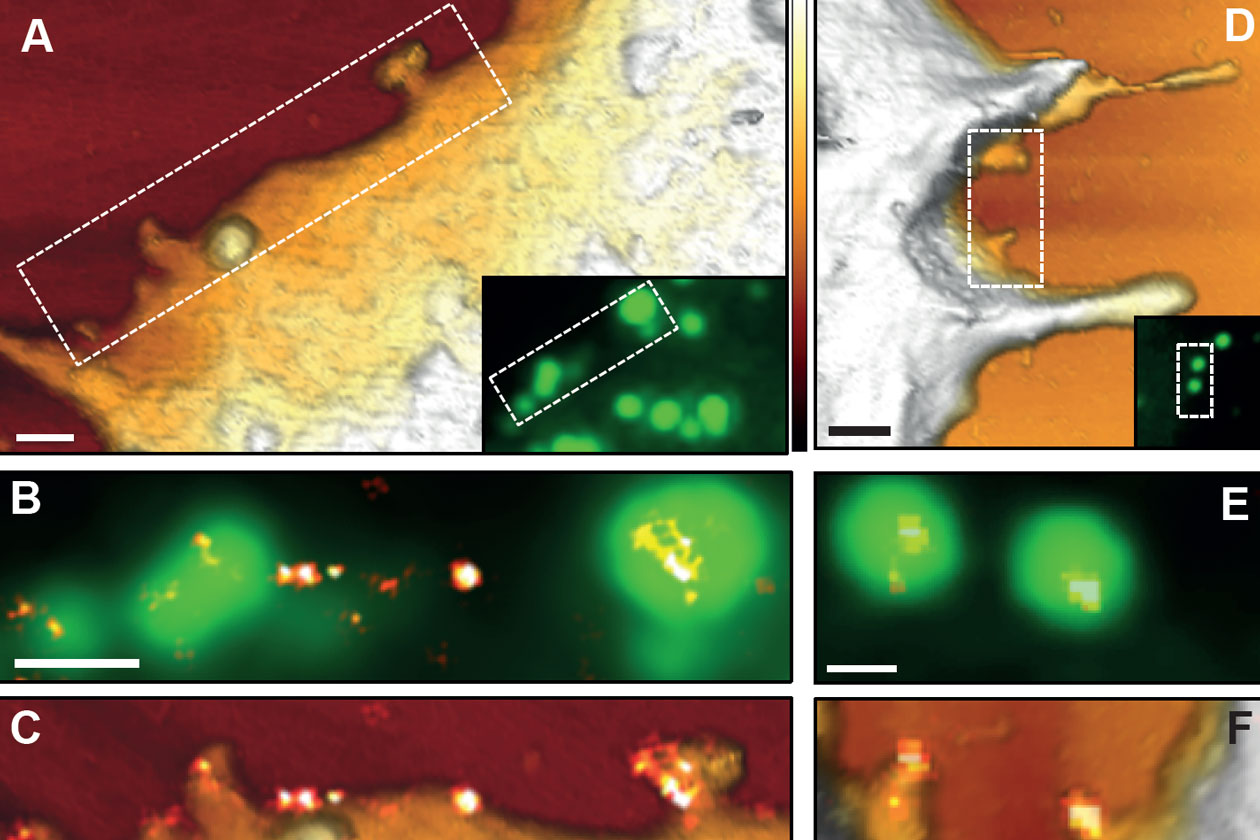
Hébergée au Centre de biochimie structurale (CBS), la plateforme PIBBS est composée de huit plateaux dédiés à la biologie structurale intégrative. Ces plateaux sont orientés sur la modélisation de macromolécules et de complexes protéine-ligand, la chemo-informatique, la radiocristallographie à haute résolution, la spectroscopie de RMN liquide, la cryo-microscopie et cryo-tomographie électronique, la microscopie à force atomique, la microscopie de fluorescence (smFRET et suivi de particules uniques), la spectroscopie de fluctuation de fluorescence (FCS et FCCS), l'analyse SAXS et la caractérisation biophysique (calorimétrie, dichroïsme circulaire et suivi de dénaturation thermique par fluorescence).
La spécificité et le point fort de la plateforme sont de rassembler au sein d’une même structure des méthodes de biologie structurale (RMN, RX, ME, bio et chemo-informatique) et des techniques de pointes de biophysique (AFM, microscopies optiques avancées en fluorescence). PIBBS offre une combinaison unique d'outils pour la caractérisation structurale de macromolécules biologiques, de leur dynamique et de leurs associations.
Expertises et services
- Analyse bioinformatique et développement d’outils de modélisation structurelle à homologie très basse (découpage de protéines, prévision des ligands, criblage in silico) avec une expertise en modélisation avec le logiciel AlphaFold,
- Analyse RMN pour la détermination de structures en solution et les études d'interactions protéine-protéine, protéine-ligand et acide nucléique-protéine,
- Cristallographie pour la détermination de structures à haute résolution,
- Cryo-microscopie électronique et tomographie,
- Microscopie à force atomique (AFM) combinée à la microscopie de localisation de molécules uniques et AFM haute vitesse,
- Caractérisation biophysique structurale de routine (diffusion de la lumière, dichroïsme circulaire, fluorescence standard),
- Spectroscopie de fluorescence avancée (FCS, FCCS, RICS, number and brigthness, fast-FLIM, single molecule, FRET et PIE),
- Microscopie de fluorescence avancée (PALM/STORM),
- Analyse de cinétiques enzymatiques transitoires à l'échelle de la milliseconde (détermination de Kon, Koff, Kd) par des techniques de stopped-flow, de quench-flow et de cryo-enzymologie,
- Développement d'outils de chemo-informatique pour la création et la gestion de bases de données de composés et de réactions chimiques, l'élucidation de leurs structures et la conception de médicaments.
Moyens et équipements
- Calcul : serveur de 280 cœurs et 100 To pour la bioinformatique et chimio-informatique, et serveur de 248 cœurs, 10 GPU et 500 To pour la microscopie électronique,
- Résonance magnétique nucléaire (RMN) : spectromètres 800 MHz et 700 MHz (Bruker) avec cryo-sondes et spectromètre 600 MHz (Bruker) avec sonde haute pression,
- Cristallographie : STARlet (Hamilton), Rock Imager 1000 (Formulatrix), Mosquito Nanodispenser et DragonFly (SPT Labtech) et Opentrons OT-2,
- Microscopie électronique : microscope 2200FS 200 kV (Jeol) avec filtre d’énergie, CP3 Plunge (Gatan), Digital Micrograph, SerialEM et Vitrobot (FEI), camera K3 à détection directe (Gatan), porte objet Elsa (Ametek-Gatan) et microscope 1400 Flash (Jeol),
- Microscopie de force atomique : 2 microscopes Nano multimodes (Bruker) avec contrôleurs Nanoscope IIIa ou VIII, 2 microscopes JPK couplés pour la détection de molécules uniques, prototype HS-AFM (Toshio Ando) et X-AFM,
- Microscopie et spectroscopie de fluorescence avancées : FCS 2 photons, smFRET TCSPC, 2 photons STED, PALM, TIRF/MFM, AxioImager (Zeiss), Anton Paar MCR 500 couplé FCS,
- Caractérisation : ITC/DSC MicroCal et MicroCal PEAK-ITC, appareil de diffusion de dichroïsme circulaire Chirascan (Applied Photophysics), lecteurs de plaque Safire 2 (Tecan) et LB940 (Berthold), appareil de diffusion de la lumière DynaPro NanoStar II (Malvern), lecteur de gel Typhoon FLA 9500, lecteurs de plaque CLARIOstar et Synergy H1, Tycho NT.6 (NanoTemper), qPCR Stratagene MX3005P (Agilent), système stopped-flow SF-61DX2 (KinetAsyst), QFM-400 (BioLogic), système de cryo-quench-flow home made.

Comment soumettre un projet ?
Pour soumettre un projet à PIBBS, vous pouvez envoyer un mail à la responsable scientifique de la plateforme, Karine de Guilen, ou aux responsables techniques des différents plateaux. La plateforme est ouverte aux chercheurs académiques et aux entreprises. Les utilisateurs peuvent utiliser les équipements de façon autonome et payante. Dans la pratique, la plupart des projets traités au sein de la plateforme sont complexes et au long cours. Ils prennent ainsi souvent la forme d’une coopération scientifique, impliquant l’expertise complémentaire de nombreux chercheurs et ingénieurs hautement qualifiés.
Exemple de projet
Etude des protéines d'interaction RNAP chez M. tuberculosis, agent de la tuberculose
Le développement de nouvelles thérapies contre la tuberculose nécessite la connaissance des mécanismes régulateurs spécifiques de l'expression des gènes permettant à M. tuberculosis (Mtb) de survivre à un traitement antibiotique et de provoquer une infection récurrente. L'équipe de Konstantin Brodolin à l’Institut de recherche en infectiologie de Montpellier (IRIM) a montré que l'initiation de la transcription de Mtb diffère de celle de la bactérie paradigme, E. coli (EcoRNAP), car elle nécessite une protéine de liaison (RbpA) à l’ARN polymérase (RNAP).
PIBBS a utilisé le transfert d’énergie de résonance de Förster (smFRET) sur molécules uniques pour démontrer que les sous-unités des facteurs d’initiation de la transcription existent sous la forme d’états ouverts et fermés, même après liaison à l’ARN polymérase chez E. Coli. Dans le cas de Mtb, la conformation ouverte est uniquement présente après liaison à la protéine activatrice RbpA et interaction avec l'ADN promoteur. Ces résultats révèlent que la dynamique conformationnelle de la sous-unité de l'holoenzyme RNAP constitue une cible pour la régulation par des facteurs de transcription et joue un rôle essentiel dans la reconnaissance du promoteur.
La collaboration avec PIBBS a par ailleurs permis de montrer que le facteur σB de réponse au stress du pathogène humain Mtb induit l'oligomérisation de l'holoenzyme RNAP en un complexe supramoléculaire composé de huit unités RNAP, dont la structure a été résolue par cryo-microscopie électronique. La formation du complexe sous la forme fermée a été suivie par microscopie de fluorescence en temps réel à l'aide d'un spectrophotomètre à flux stoppé SF-61 DX2. L’ensemble des résultats démontre que l'activateur transcriptionnel RbpA empêche la formation d'octamères en favorisant la conformation de la RNAP fermée compatible avec l'initiation. Une région non conservée de σ constitue donc un contrôleur allostérique de l'initiation de la transcription. Le travail réalisé a enfin permis d'établir comment les facteurs de transcription basaux peuvent réguler l'expression des gènes en modulant l'assemblage et l'hibernation de l'holoenzyme RNAP.
Pour en savoir plus : Morichaud Z. et al. (2022). Structural basis of the mycobacterial stress-response RNA polymerase auto-inhibition via oligomerization. Nature Communications, 14(1):484.
Contact
PIBBS
29 rue de Navacelles
34090 Montpellier
Région : Occitanie +33 (0)4 67 41 77 01
+33 (0)4 67 41 77 01
karine.deguillen@cbs.cnrs.fr
Site de la plateforme
THÉMATIQUES : Bioinformatique, bases de données, Biologie structurale, biophysique, biochimie, Imagerie cellulaire
RESPONSABLES SCIENTIFIQUES :
Karine de Guillen
TUTELLES : CNRS, Inserm, Université de Montpellier
INFRASTRUCTURES NATIONALES : France-BioImaging, FRISBI
LABELLISATION IBiSA : 2008
MOTS CLÉS : Biologie structurale, Biophysique, Imagerie, Macromolécules, Interactions moléculaires, Haute résolution, Molecule unique, Ligand, Criblage, Tweezers, RMN
Fiche mise à jour en 2025










