Toulouse réseau imagerie (TRI)
Visualisation et analyse des fonctions biologiques animales ou végétales, de l’échelle nanométrique à l’organisme entier, par microscopie photonique, électronique, cytométrie et tri cellulaire.

Toulouse réseau imagerie (TRI)
Visualisation et analyse des fonctions biologiques animales ou végétales, de l’échelle nanométrique à l’organisme entier, par microscopie photonique, électronique, cytométrie et tri cellulaire.
Toulouse réseau imagerie (TRI)
La plateforme TRI s’intéresse à la visualisation des fonctions biologiques, de l’échelle nanométrique à l’organisme entier, avec quatre champs de compétences : la microscopie photonique, la microscopie électronique, la cytométrie - tri cellulaire et l'analyse des données associées. Elle fédère seize plateaux d’imagerie répartis sur neuf sites toulousains et accueille annuellement 800 chercheurs et étudiants appartenant à 180 équipes de recherche et 22 laboratoires privés.
Les domaines d’expertises de la plateforme TRI dans les techniques d’imagerie et cytométrie - tri cellulaire sont, d’une part la biologie animale (bactérie, levure, Homme, drosophile, abeille, poisson, amphibien, poulet, souris, rat, mouton, singe), et d’autre part la biologie végétale (plantes et microbiotes, interactions plantes-microorganismes et développement).
Pour l’ensemble de ces modèles, les observations sont réalisées sur des cultures cellulaires, des préparations tissulaires, des embryons, des organes, des animaux entiers ou des plantules. 44 personnels dédiés, ingénieurs et techniciens, mettent en œuvre leurs compétences et expertises sur des technologies extrêmement vastes et de très haut niveau couvrant l’imagerie cellulaire et tissulaire in vivo, la microscopie de super-résolution , la cytométrie, l’imagerie moyen et haut débit, la microscopie électronique en transmission ou à balayage et les cryométhodes, la microscopie à force atomique, l’imagerie des interactions moléculaires, l’imagerie du petit animal, le traitement des données et la modélisation.
Expertises et services
Microscopie photonique :
- Macroscopie, microscopie plein champ, confocale mono et biphotonique, macroscopie et microscopie à feuille de lumière sur cellules, tissus et petits organismes vivants,
- Vidéomicroscopie et bioluminescence intravitale et corps entier sur le petit animal,
- Imagerie à haut contenu et haut débit (Opera et Operetta),
- Microscopie confocale rapide pour l’analyse de cellules infectées par le VIH et le bacille de la tuberculose en confinement A3,
- Microscopie de super-résolution PALM, STORM, STED et SIM,
- Microscopie à force atomique (AFM),
- Microscopie vibrationnelle CARS/SRS.
Microscopie électronique :
- Préparation d’échantillons pour observation en MET ou MEB, y compris avec des cryométhodes (cryofixation, cryosubstitution, technique CEMOVIS),
- Acquisition d’images, analyse et interprétations des données en MET (tomographie et spectrométrie par perte d’énergie), MEB (analyse X) et cryo-EM.
Cytométrie et tri cellulaire :
- Cytométrie, cytométrie spectrale, cyto-imagerie et tri cellulaire en confinement A2,
- Cytométrie et tri cellulaire sur cellules infectées par le VIH et la tuberculose en milieur A3.
Analyse de données :
- Analyse et interprétation d'images en microscopie photonique et électronique,
- Analyse et interprétation de données en cytométrie.
Moyens et équipements
Microscopie photonique :
- 5 macroscopes Zeiss, Leica et Olympus,
- 12 microscopes plein champ Zeiss, Leica et Nikon,
- 3 microscope à fauille de lumière,
- 5 spinning disk PerkinElmer et Nikon, dont un en zone A3,
- 10 microscopes confocaux monophotons et 5 biphotons, dont un en zone A3,
- 3 systèmes TIRF,
- Système de pinces optique,
- 5 microscopes de super-résolution PALM, dSTORM et SIM,
- 4 systèmes de mesure de durée de vie de fluorescence avec 2 TCSPC et 2 streak cameras,
- 2 microscopes à force atomique (AFM),
- 4 scanners de lames,
- 3 systèmes d’imagerie à haut débit Opera et Operetta,
- Microscope Raman CARS/SRS.
Microscopie électronique :
- 4 systèmes de coupe ultrafine,
- 3 cryofixateurs et 3 cryosubstituteurs,
- 5 microscopes électronique en transmission (MET) et à balayage (MEB).
Cytométrie et tri cellulaire :
- 11 cytomètres,
- 5 trieurs,
- 2 cyto-imageurs.
Traitement d’images et modélisation :
- 15 postes de traitement et d’analyse d’images,
- Logiciels de reconstruction 3D, logiciels de mesure et de quantification 2D et 3D, logiciels d'analyse de cytométrie et de chimiométrie.
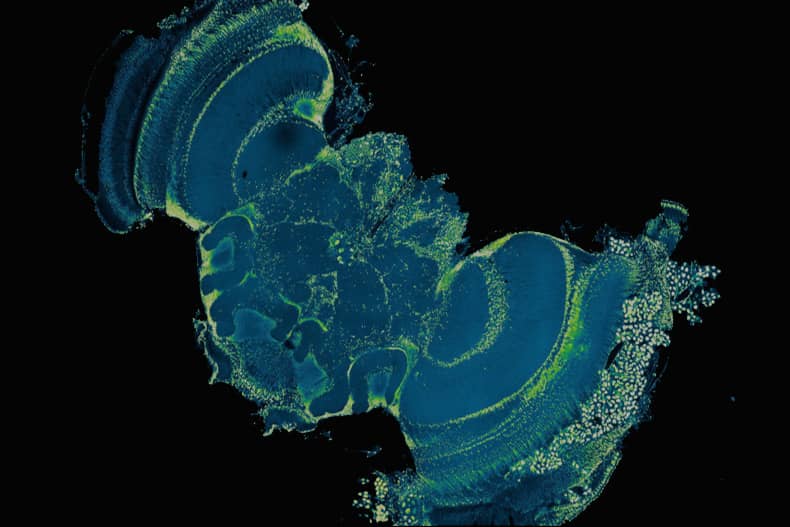
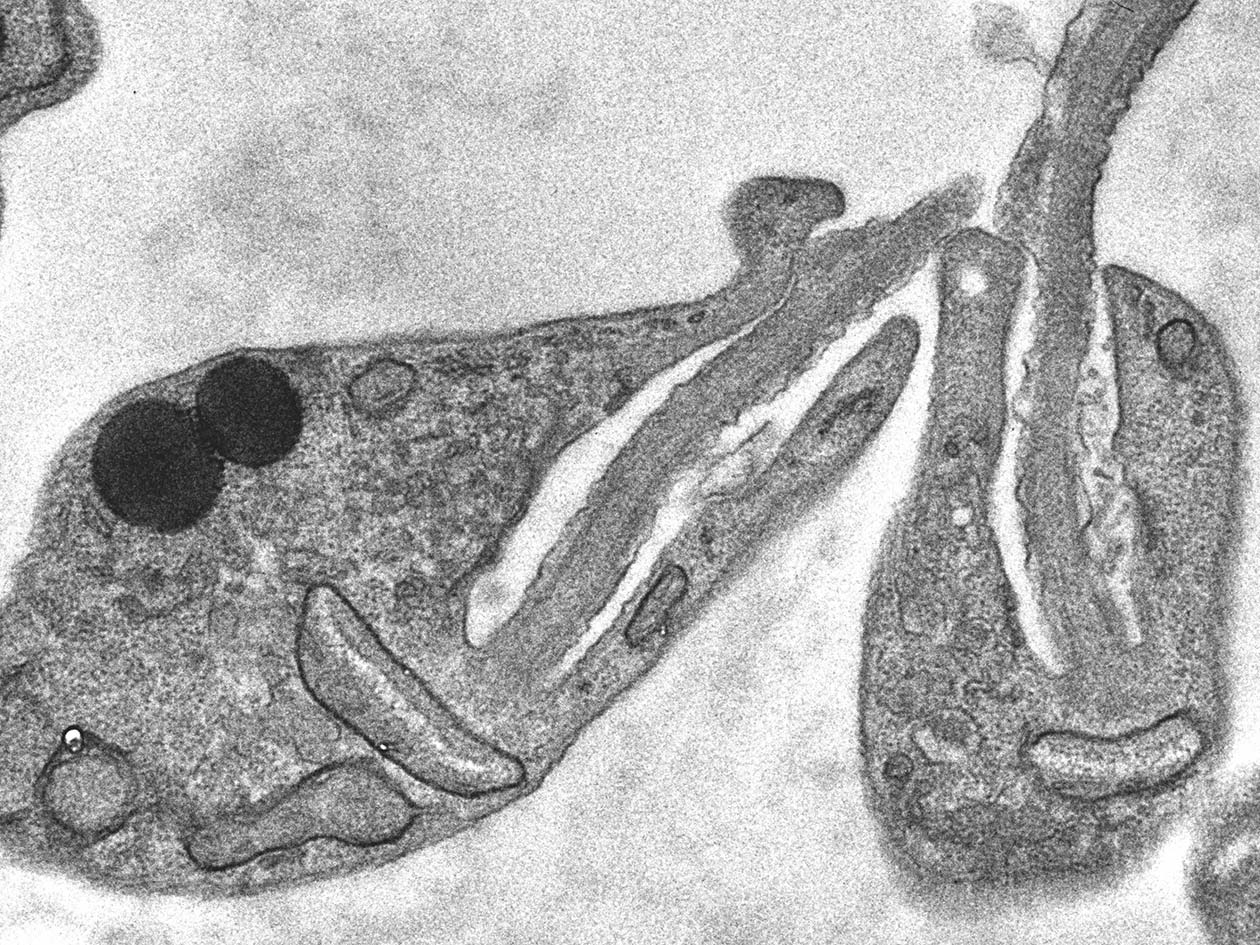

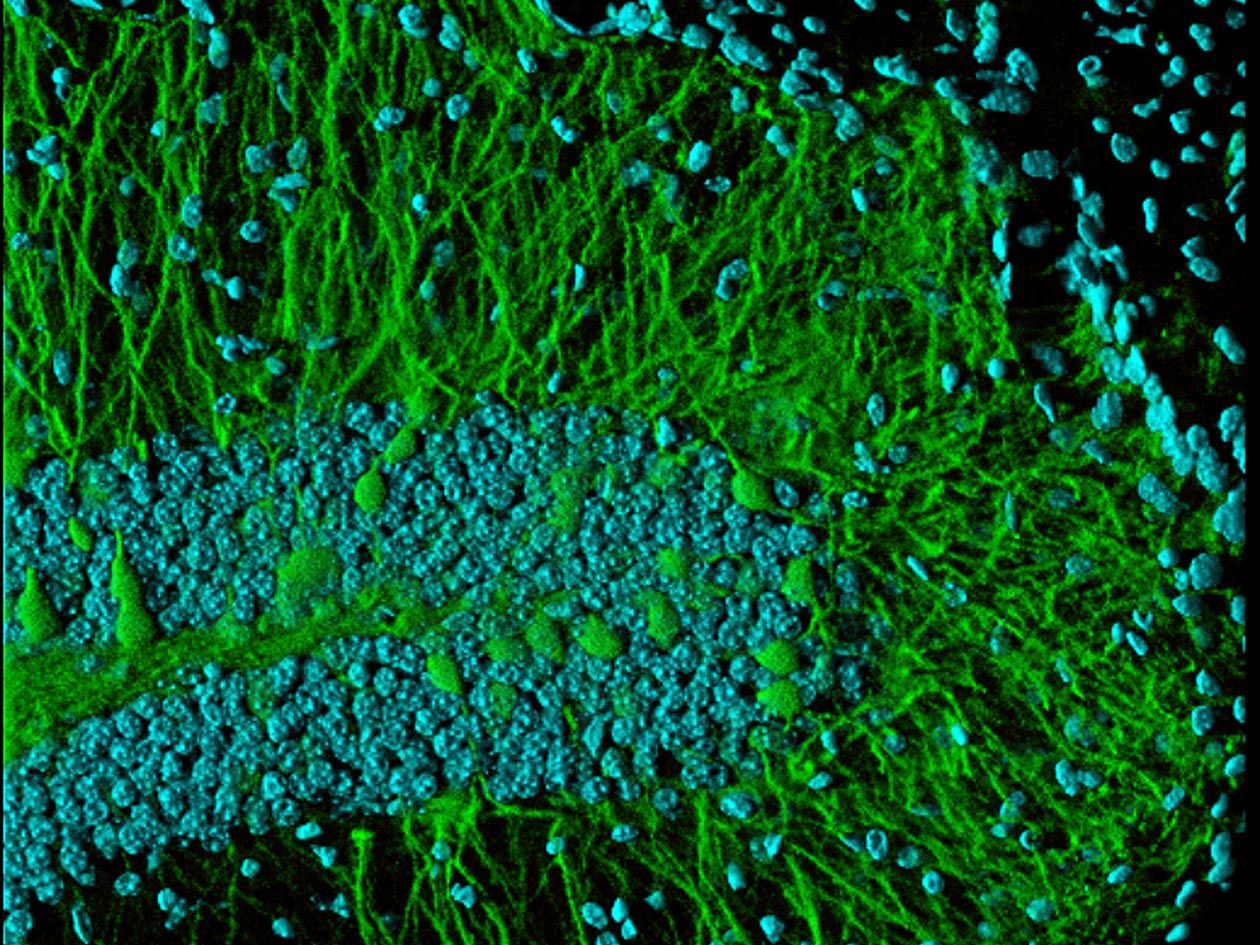
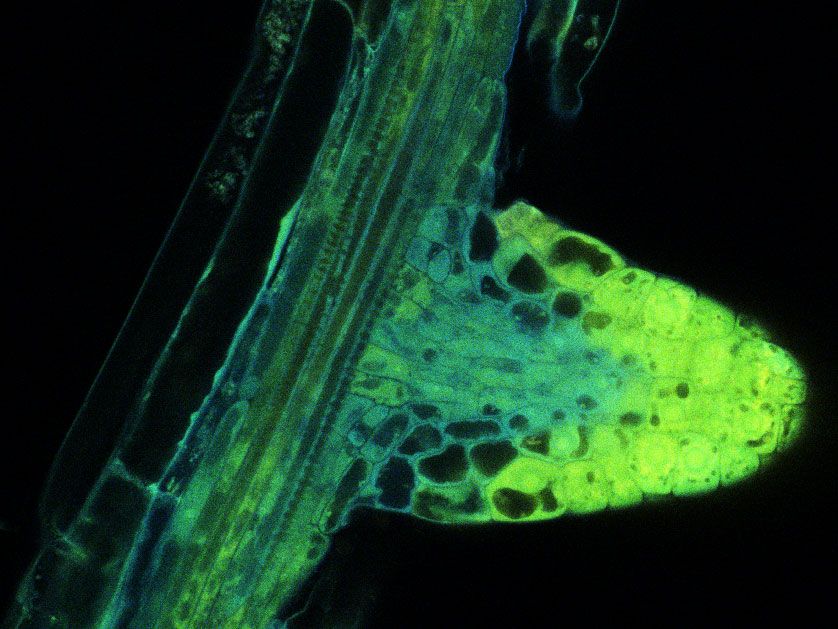

Comment soumettre un projet ?
Point d’entrée unique, le site internet recense l’ensemble des domaines, expertises et plateaux de la plateforme TRI. Pour toute demande d’information, de prestation ou de projet, envoyez un mail à l'adresse direction@genotoul.fr. Vous serez orienté vers les experts et les technologies les plus à même de répondre à votre besoin. Le responsable du plateau concerné vous recontactera ensuite pour discuter plus précisément de votre projet et vous faire remplir une fiche formalisant la demande (projet, équipements nécessaire, type de prestation…).
Exemple de projet
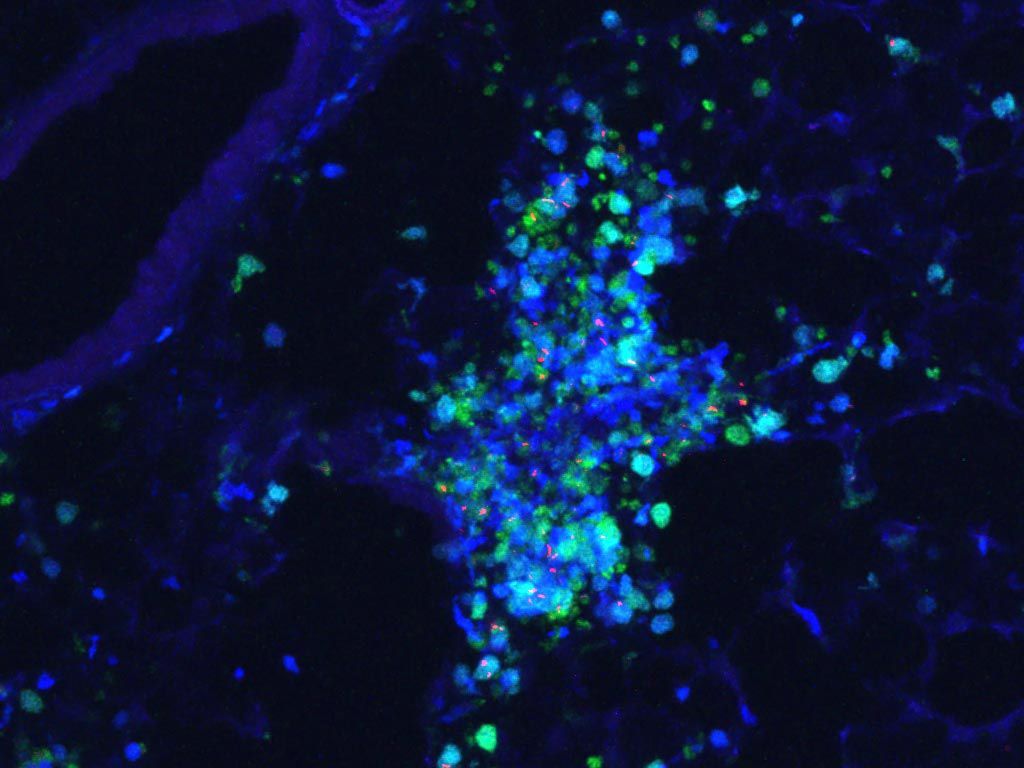
Découverte du compartiment chromatien D induit par les lésions de l’ADN
En étudiant la réparation des cassures double brin et l’organisation tri-dimensionnelle de notre génome, notamment grâce aux techniques de FRAP et de super-résolution (3D RIM) disponibles sur la plateforme TRI, l’équipe de Gaelle Legube au Centre de biologie intégrative (CBI) de Toulouse a démontré qu'à la suite de cassures, notre ADN subit des changements organisationnels de structure. En particulier, les cassures double brins conduisent à la formation d’un nouveau compartiment chromatinien appelé « compartiment D ».
Ce compartiment D contient différentes cassures qui se regroupent au même endroit, ce qui favorise l’apparition de translocations aux effets néfastes pour la stabilité du génome. Cependant, ce compartiment rassemble également certains gènes qui jouent un rôle primordial dans la signalisation à la cellule de la présence de dommages. La présence de ces gènes dans ce compartiment faciliterait leur activation, permettant ainsi à la cellule de répondre efficacement aux menaces posées par les cassures double brin de l’ADN. Ainsi, la cellule aurait un avantage sélectif à maintenir ce compartiment D.
Ces travaux éclairent notre compréhension de l’impact de la réorganisation tri-dimensionnelle de notre ADN sur les mécanismes que la cellule met en place pour faire face au danger causé par les cassures double brin de l’ADN. Mais le revers de la médaille est que cette réorganisation peut occasionnellement entrainer des effets néfastes pour la cellule en générant des translocations chromosomiques, qui sont des événements délétères à l’origine de cancers.
Pour en savoir plus : Arnould C. et al. (2023). Chromatin compartmentalization regulates the response to DNA damage. Nature, 623:183-192.
Contact
Toulouse réseau imagerie (TRI)
LBME, CNRS, Bâtiment IBCG
Université de Toulouse
118 route de Narbonne
31062 Toulouse
Région : Occitanie +33 (0)5 61 33 59 39
+33 (0)5 61 33 59 39
direction@genotoul.fr
Site de la plateforme
THÉMATIQUES : Cytométrie, Imagerie cellulaire, Imagerie in vivo, radiobiologie
RESPONSABLES SCIENTIFIQUES :
Olivier Gadal
RESPONSABLES TECHNIQUES :
Cecile Pouzet, Jacques Rouquette
TUTELLES : CNRS, Inserm, Université de Toulouse
INFRASTRUCTURES NATIONALES : France-BioImaging
LABELLISATION IBiSA : 2009
MOTS CLÉS : Microscopie photonique, Microscopie électronique, Microscopie à force atomique, Cytométrie, Imagerie cellulaire, Imagerie tissulaire, Imagerie infectieuse, Imagerie corps entier, Imagerie, Cyto-imagerie, Tri cellulaire, Cryométhodes, F-technique, Intravital, Plante, Animal, Homme, Analyse d’image, Tomographie, Microorganismes
Fiche mise à jour en 2024












